戴源宏 0417082
Clustalw是進行多序列比對的軟體,多序列比對是指對3條以上的DNA/RNA或蛋白質序列進行序列比對,可以推導出序列的同源性以及尋找序列上具有保守性(conservation)的區域。保守區域往往是影響蛋白質功能非常重要的區域,找到保守區域並對保守區域進行修改可能可以改進蛋白質的功能或效率。多序列比對對研究很有幫助。
在Terminal輸入sudo apt-get install clustalw,並輸入密碼。
下載安裝中...
安裝完成
輸入clustalw開啟。
開啟後出現目錄:
1.加載序列文件。文件的格式可以有clustal, fasta等。
2.進行多序列比對。把加載的序列進行比對。
3.進行結果比對。這項可以用之前的比對結果比對新序列。
4.畫進化樹。將比對結果畫成樹狀圖,可以看出各序列的相似程度。
S.這項可以下達terminal的指令
H.幫助目錄。開啟幫助目錄, 對各選項進行解釋。
X.離開。
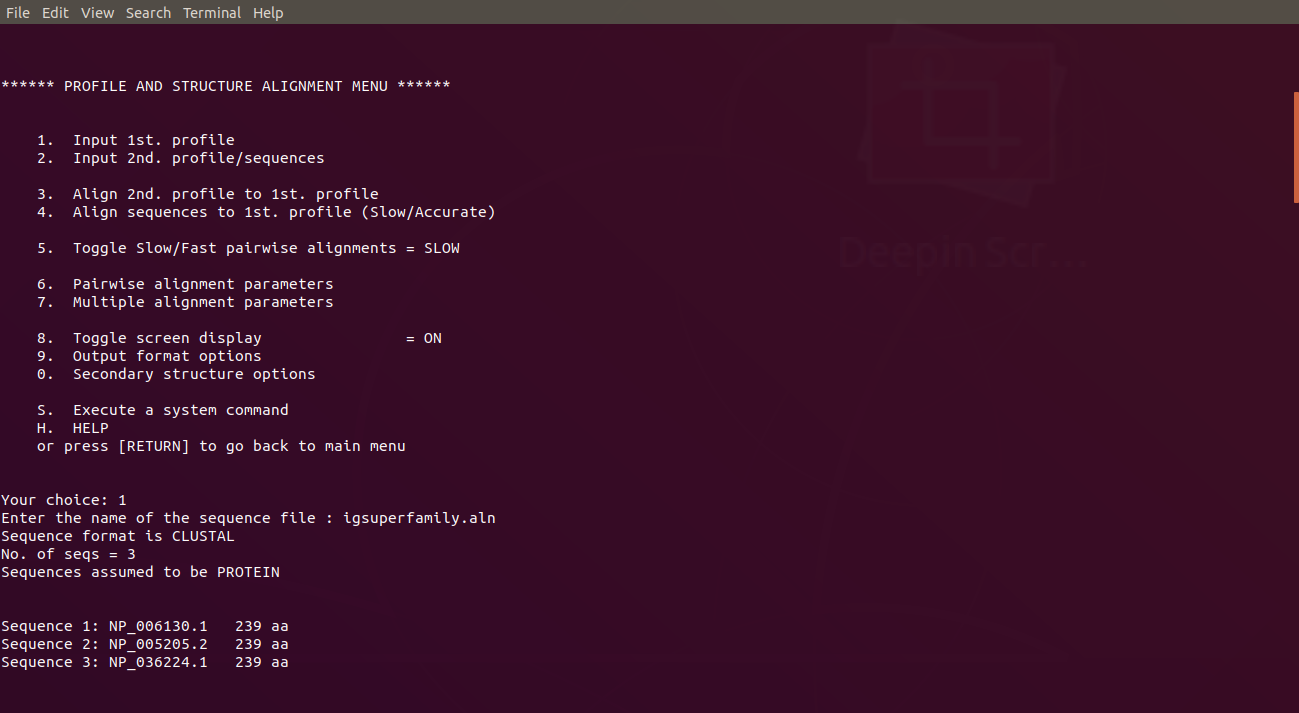
輸入1選擇加載序列文件,輸入文件名稱。注意所有要進行比對的序列要放在同個文件中。
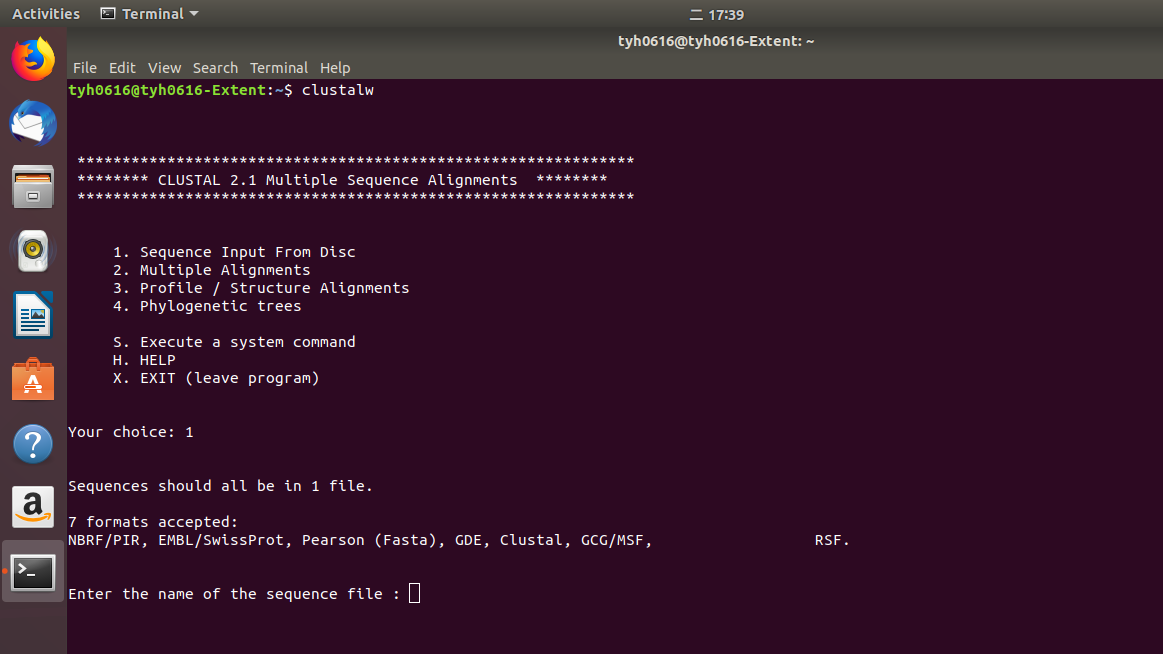
這是三條Ig Super Fanily的immunoglobulin蛋白序列,做為示範。格式為Fasta。
輸入文件名稱igsuperfamily.fas
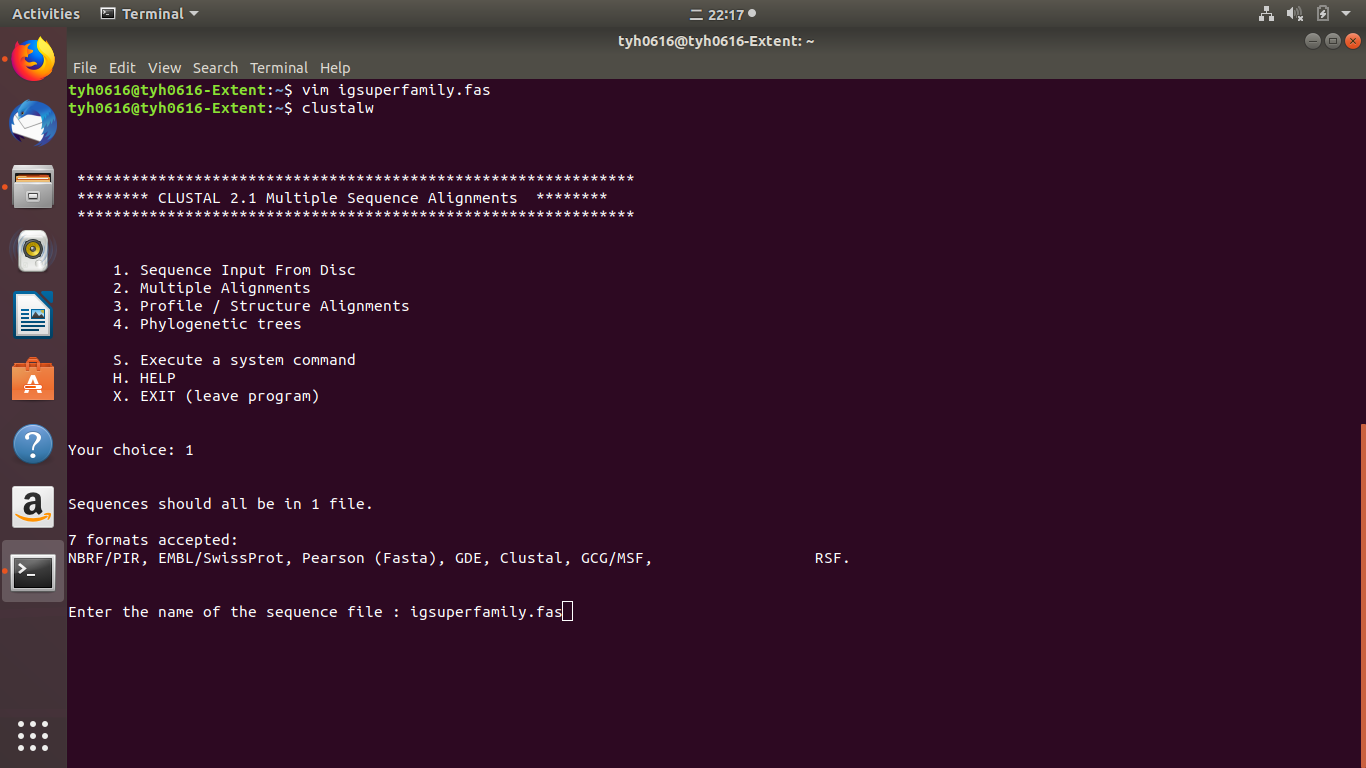
成功載入三條序列。載入後會跳回目錄。輸入2進入比對目錄
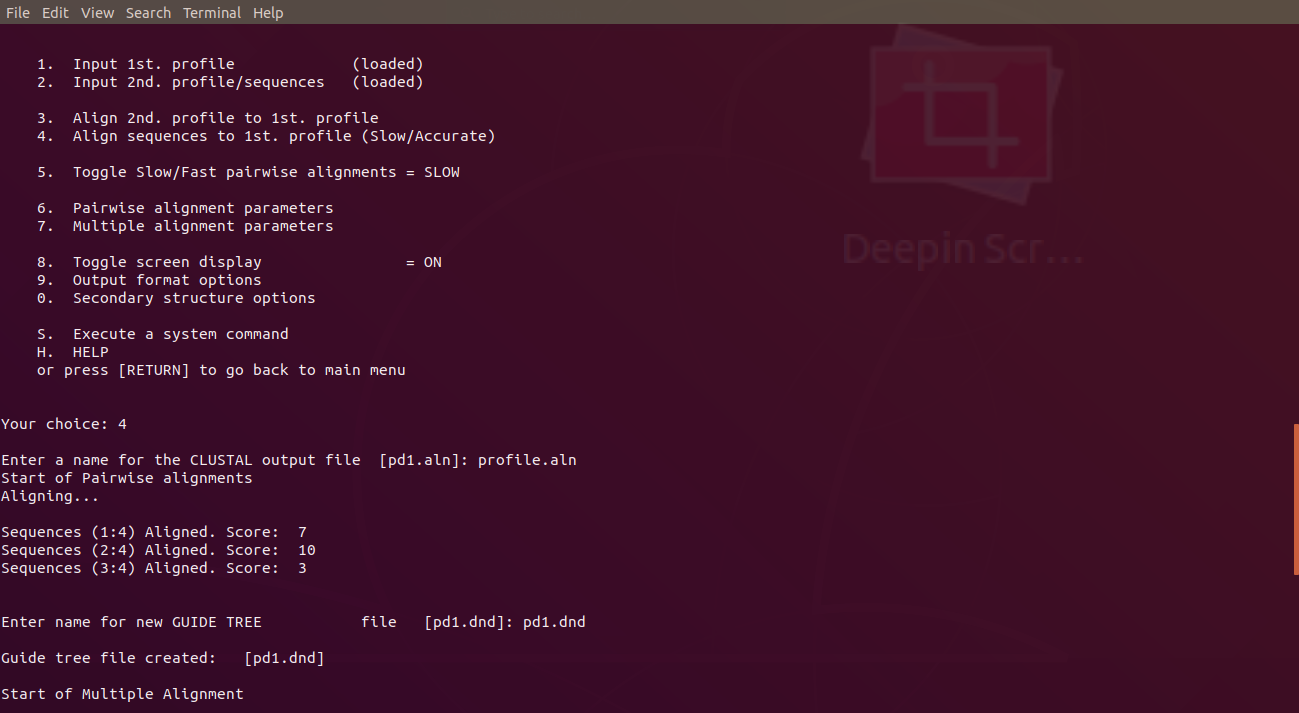
比對目錄的第一項就是進行比對。輸入1後會要求輸入比對結果文件名,輸入名字即可。
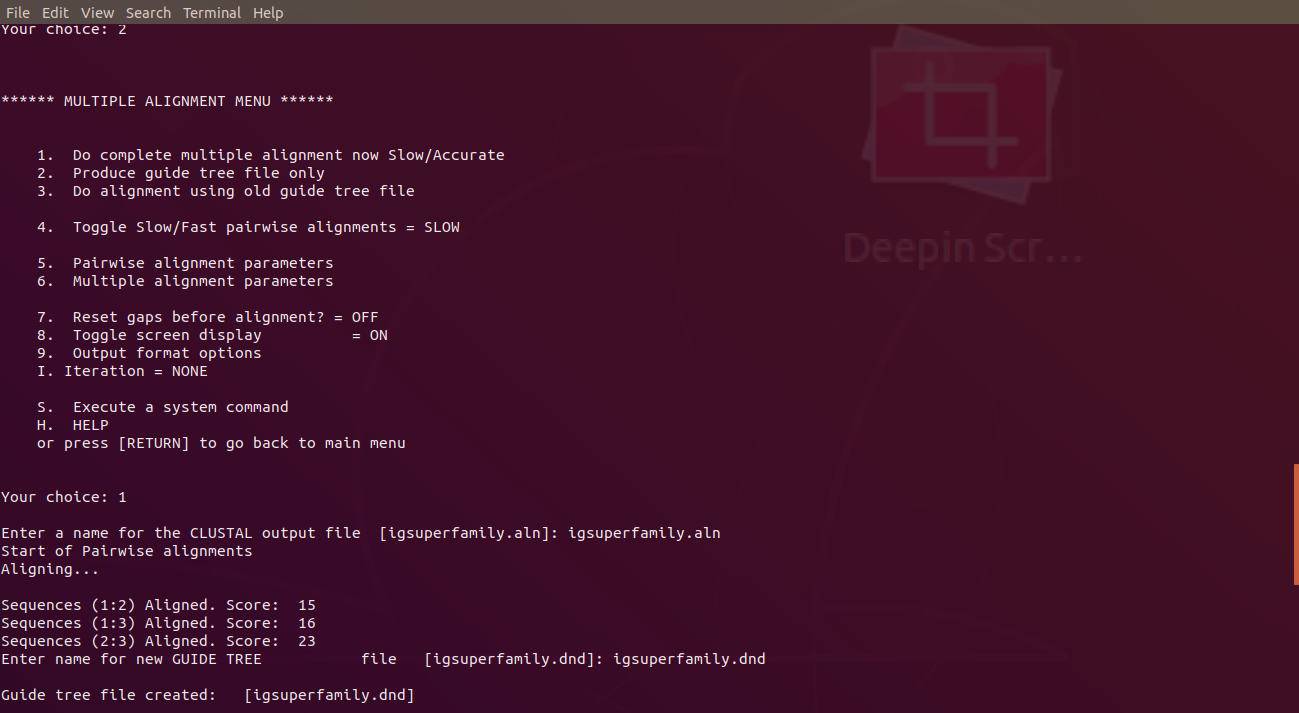
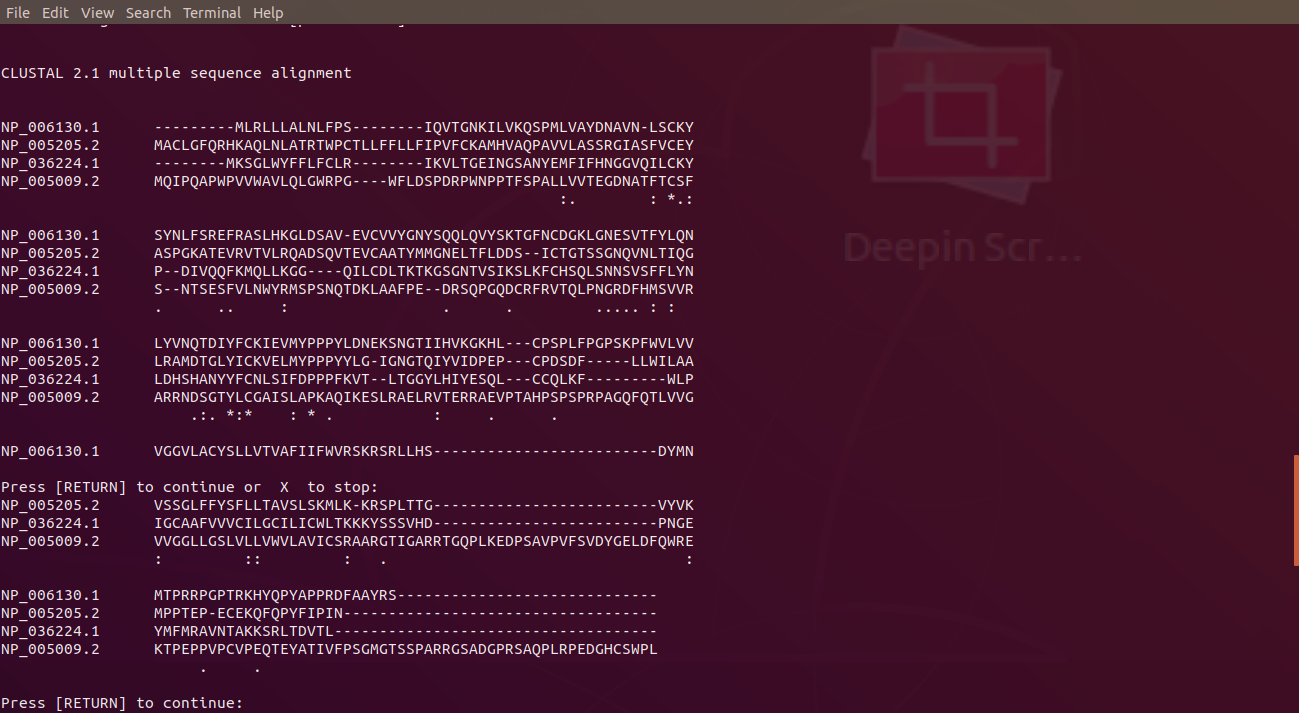
比對結果出爐,三條序列下方有點和*符號,點越多則表示這個胺基酸相同的序列越多,*符號則是所有序列在這個胺基酸都是相同的。從結果的第三行可以看到有連續三個*符號,表示這裡很可能是保守區域。
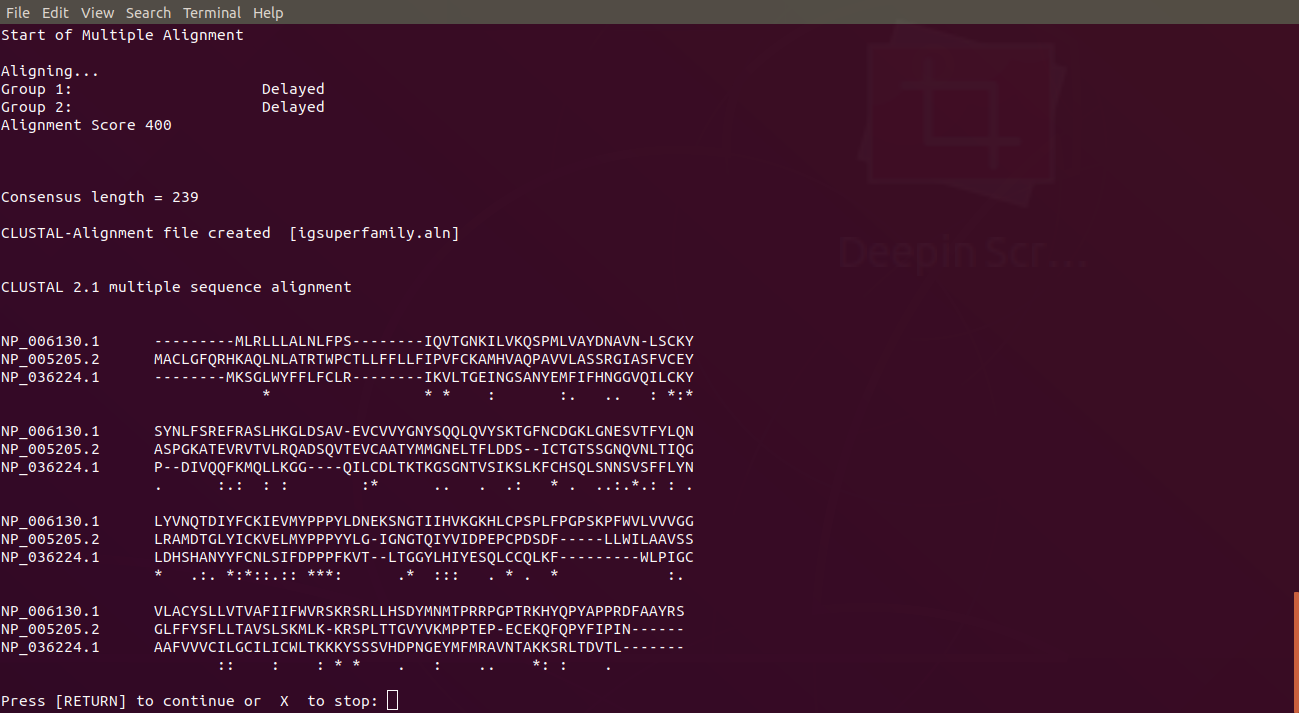
輸入3,可以用比對結果比對新的序列。
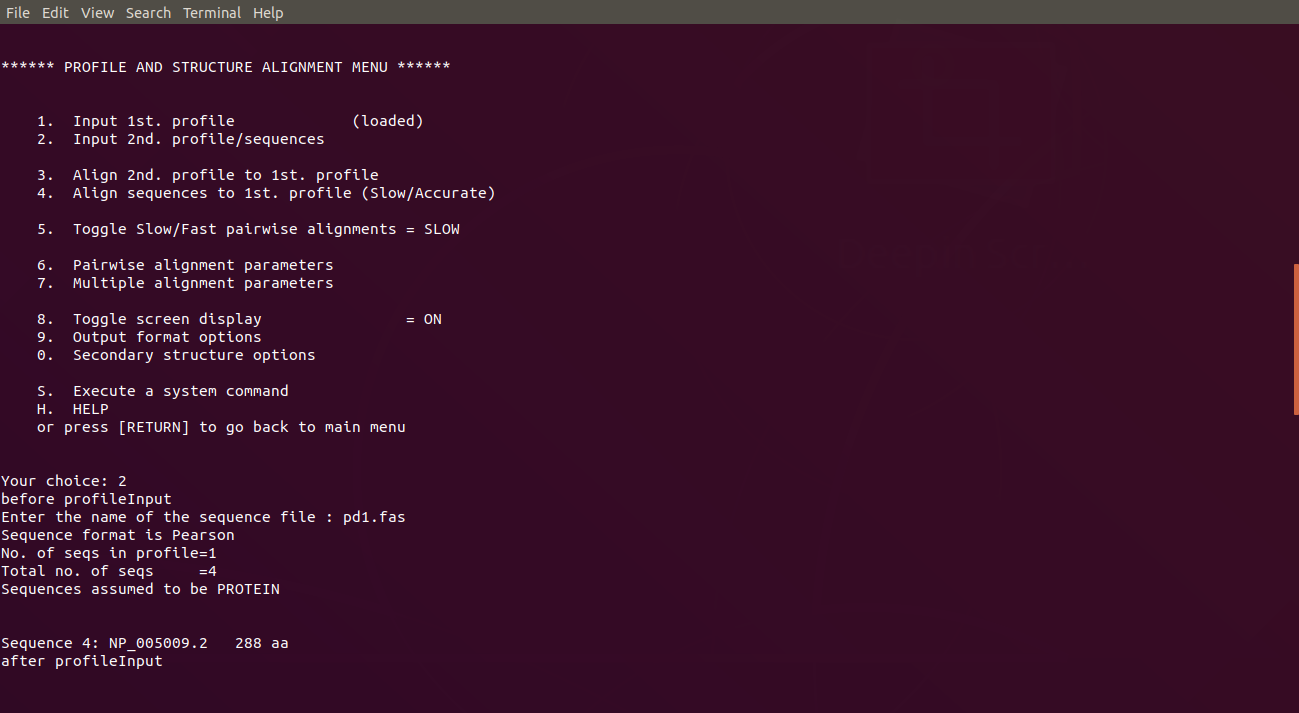
1.加載之前的比對結果(.aln文件)
2.加載新序列或另一次的比對結果。
3./4.分別進行結果對序列和結果對結果的比對。
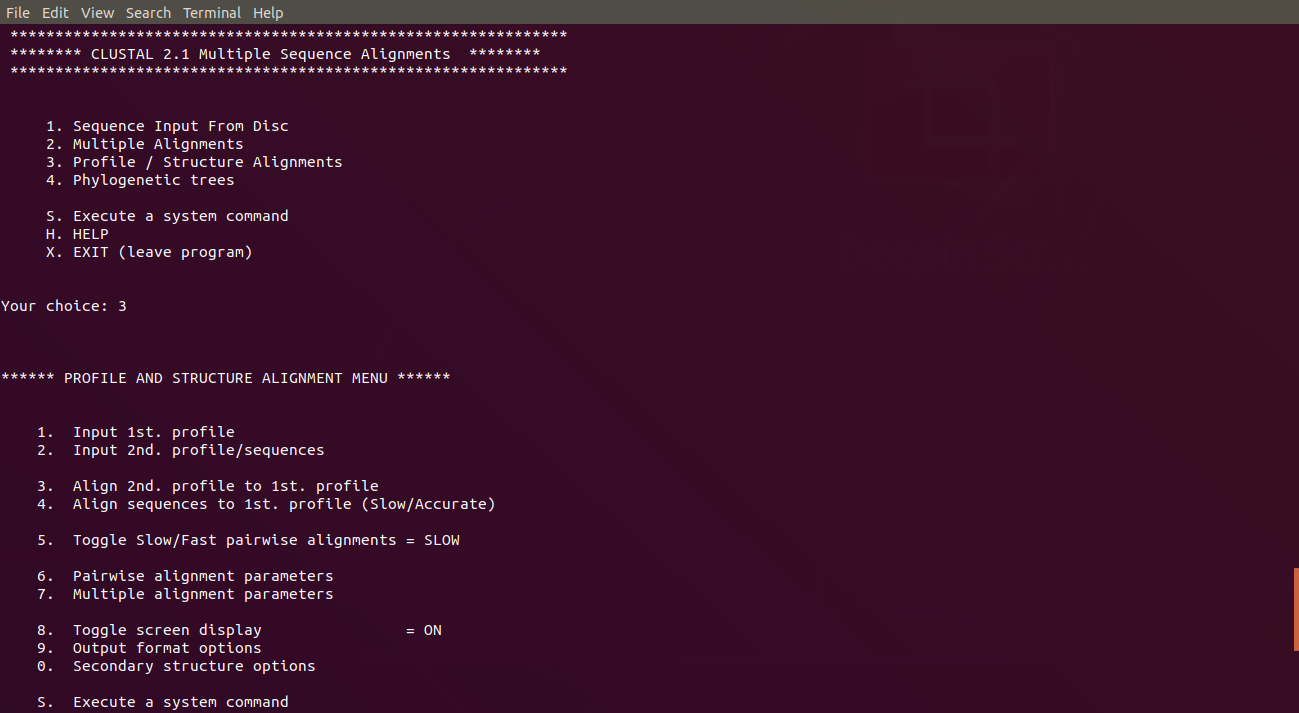
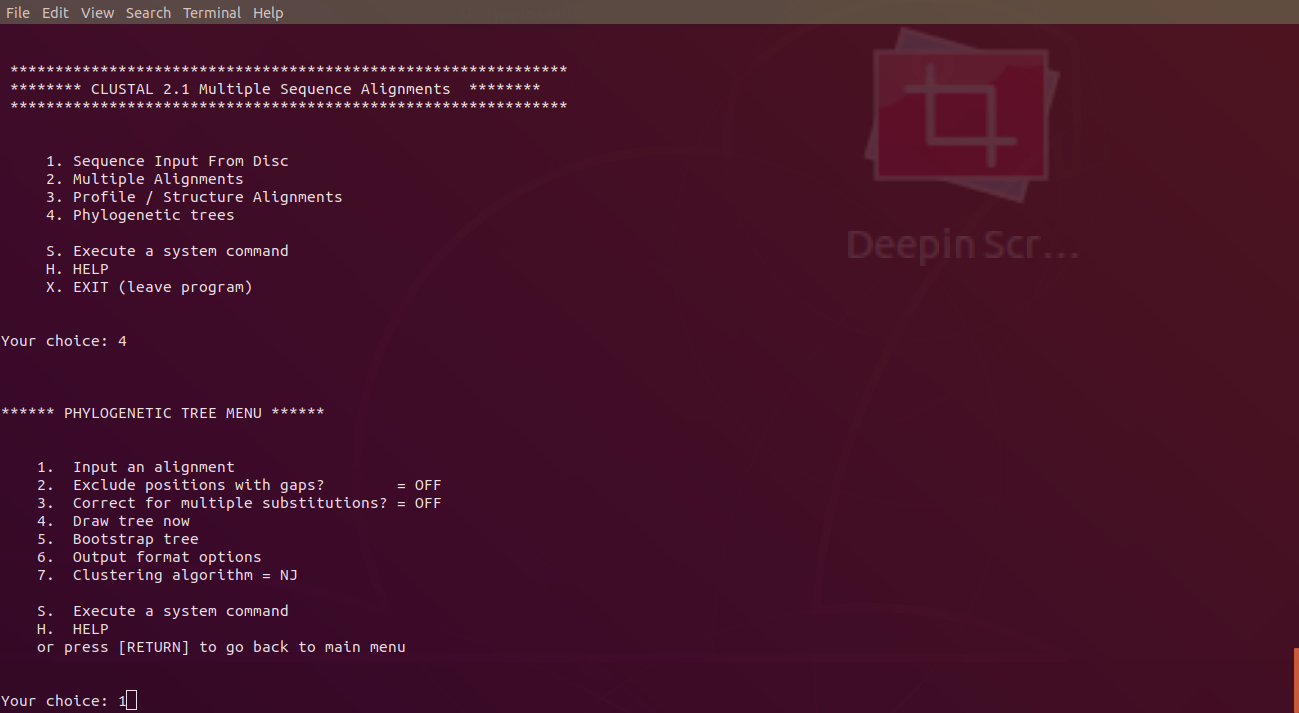
目錄的第4項,可以畫演化樹狀圖。
輸入1,加載比對結果
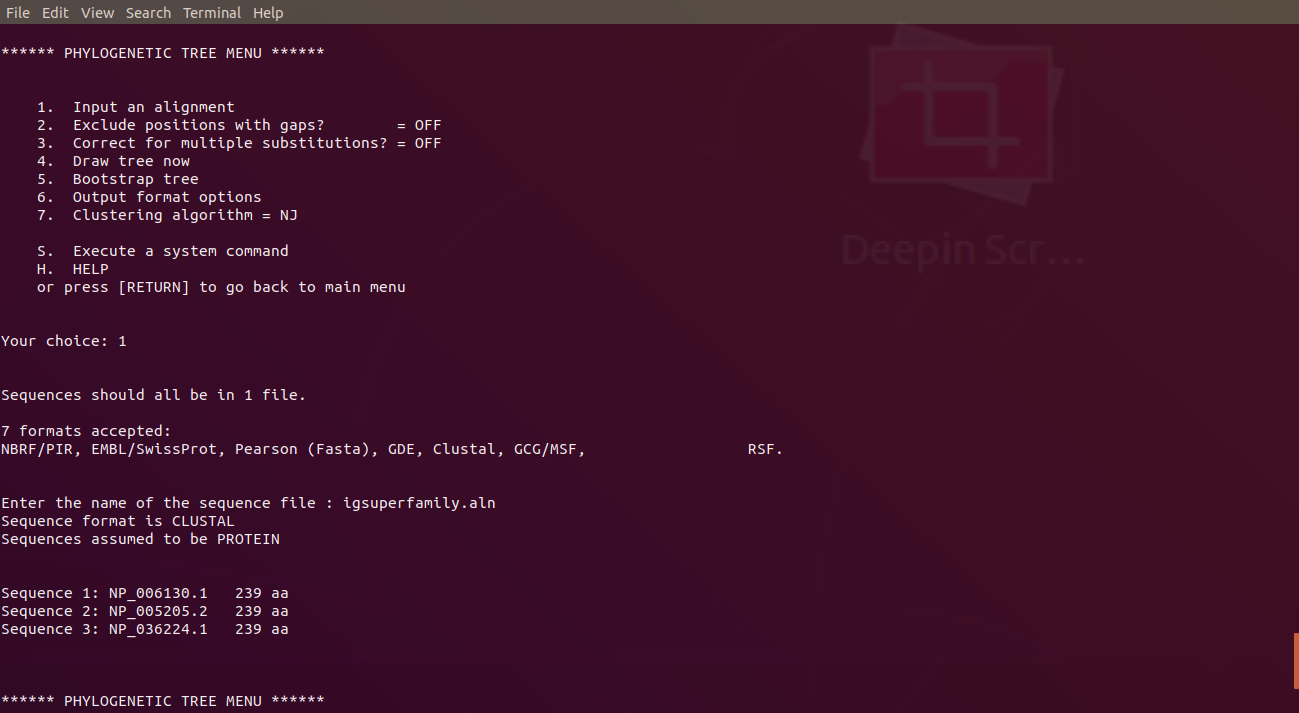
接下來輸入4,畫樹狀圖
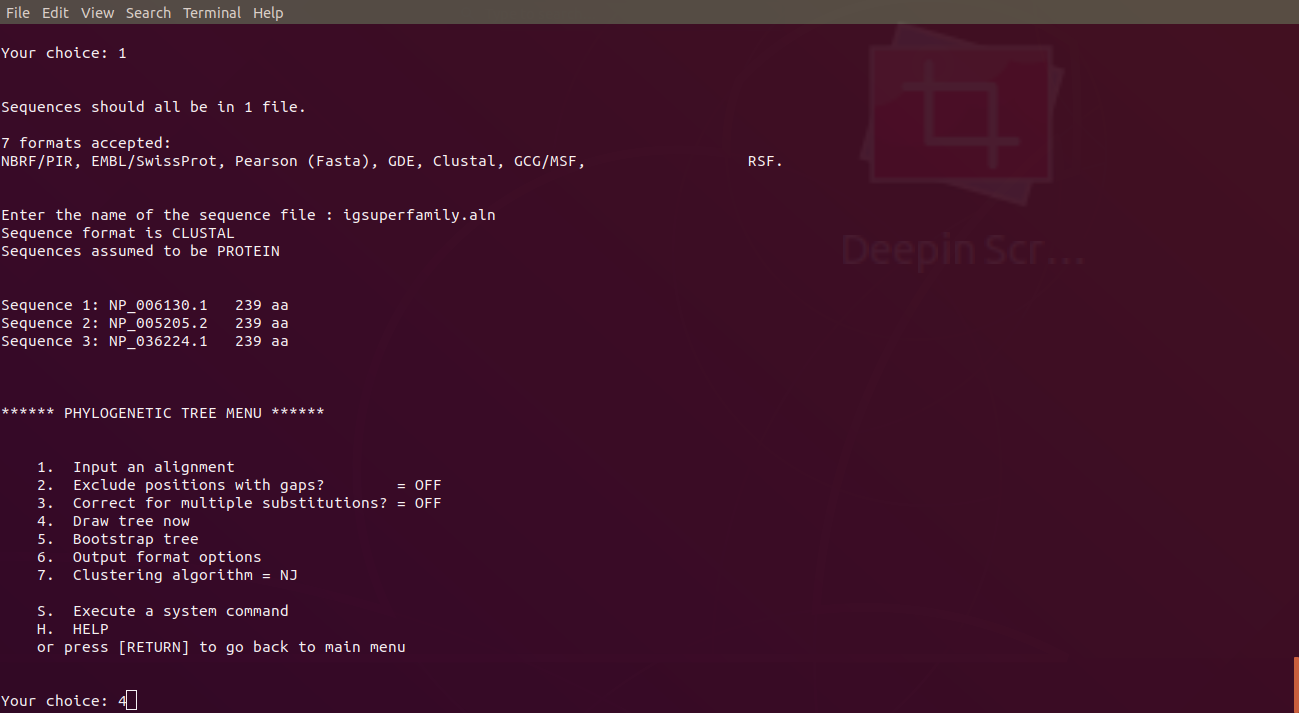
要求輸入樹狀圖的文件名。
輸入後就會給出樹狀圖的文件。
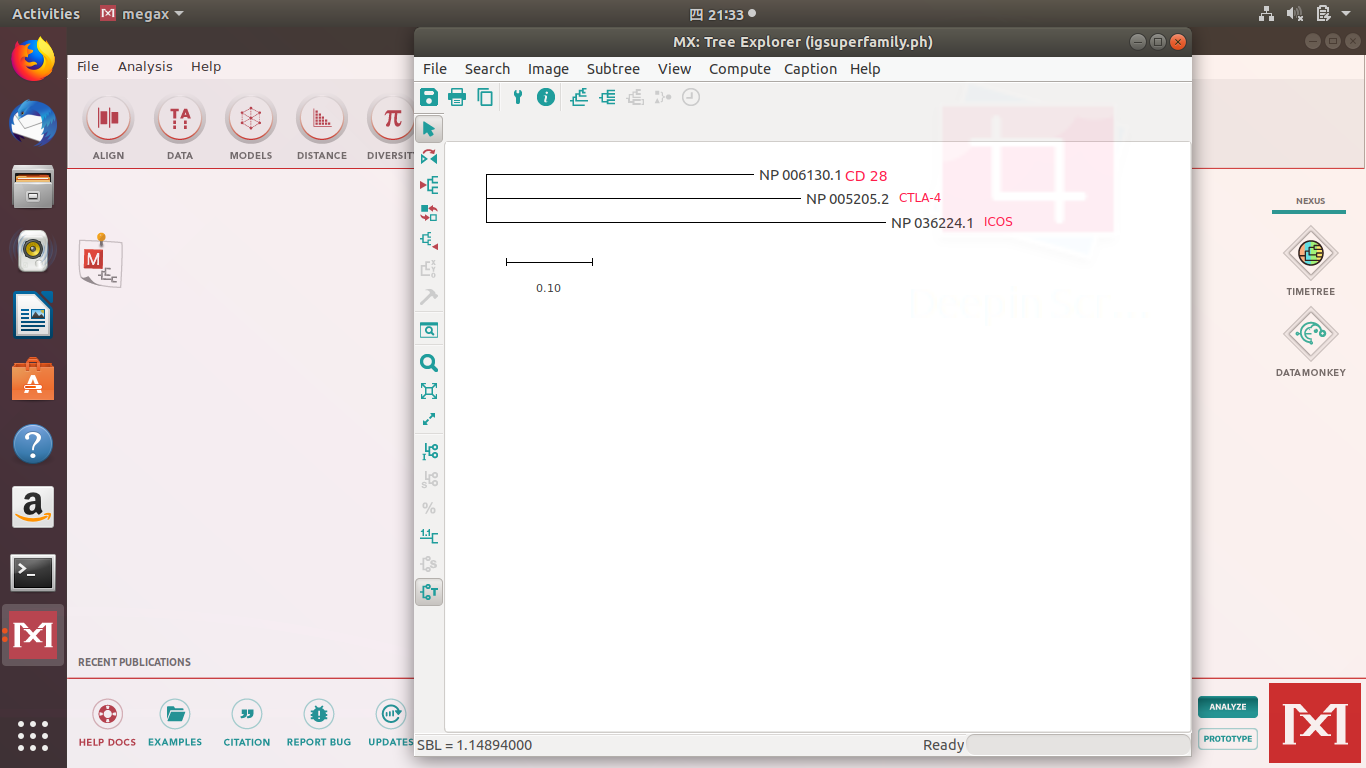
軟體內並不能顯示圖片,需要另外使用軟件開啟樹狀圖。我使用的的MegaX。
從樹狀圖可以推測CD28比較早出現,然後才是CTLA-4和ICOS。
Clustalw的使用就介紹到這裡。
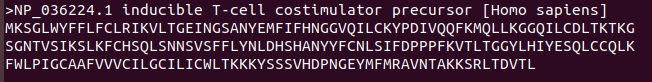
Answer: Fasta format是序列常用的格式,格式以>开头,后面接着序列名称,下一行开始DNA或氨基酸的序列,例子如下:
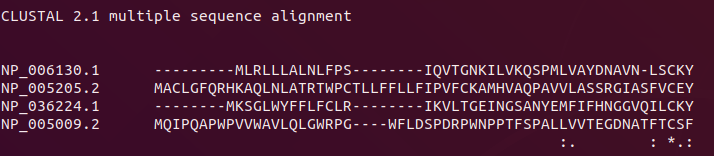
Clustalw format是在进行Clustalw序列比对时才会用到的格式,格式如下:
1.根据输入的序列数量,就会有多少行。每行以序列名称开头,空格后就是序列,中间根据比对结果会出现-符号,代表这里没有氨基酸或碱基。序列长度大于60则跳行。
2.最下行是比对结果。*符号是每个序列在这个位置的氨基酸或碱基都一样,两个点是大部分序列在这里是保守的,一个点是一半的序列是保守的。
例子:
Answer: 先对各序列进行两两对比,然后选择相似度最高的两条进行序列比对,并形成家族树。剩下的序列将会依照相似度加入家族树中进行比对。
www.clustal.org