Application of Pymol on structural biology
一. 安裝與基本操作:
在軟體中心尋找"pymol",且按install
系統會要求輸入密碼以取得權限安裝
安裝成功後在左邊快捷列會看到底下紅框的ICON

左鍵兩下便可開啟相關介面
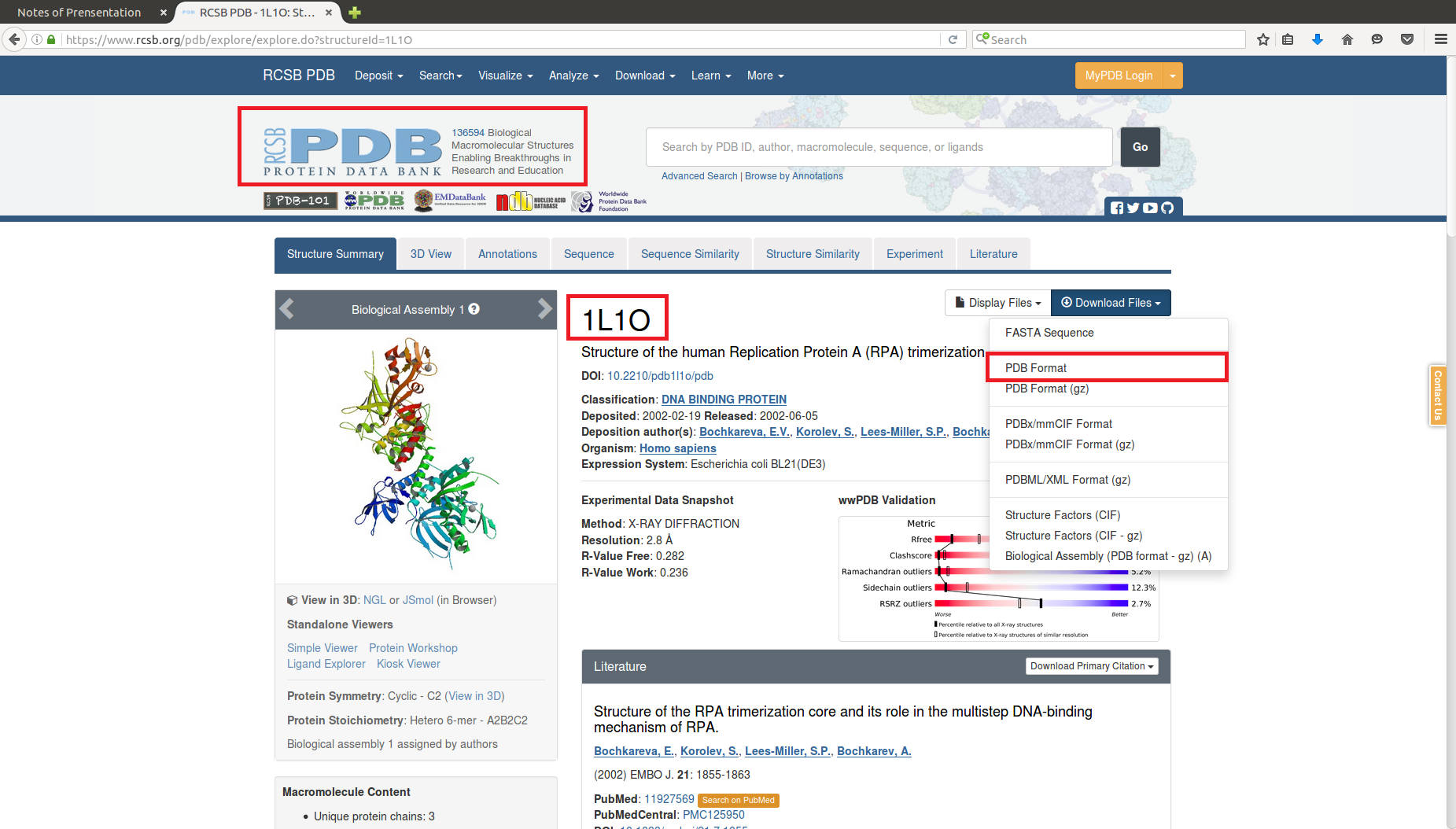
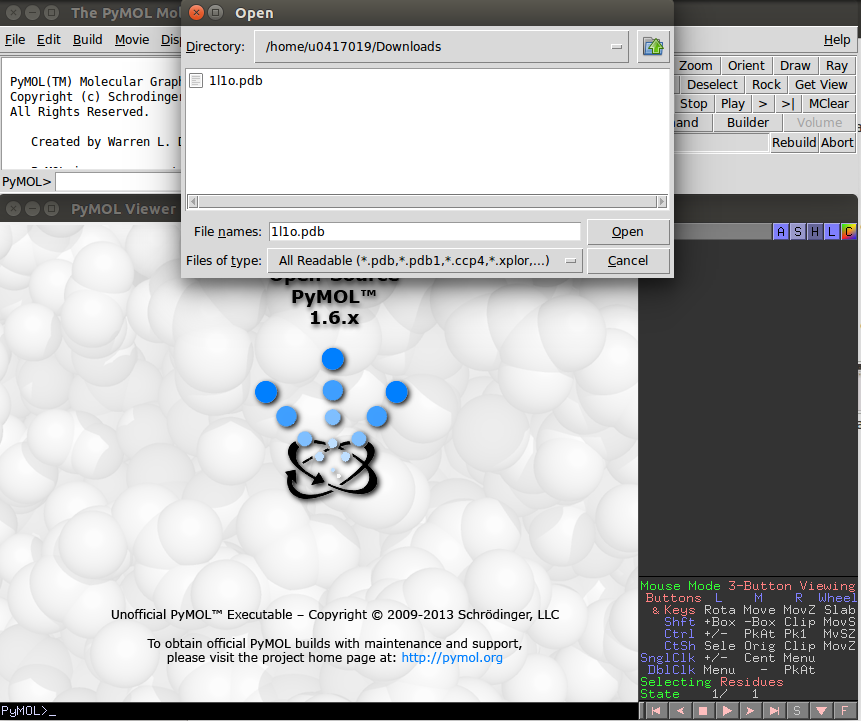
至PDB尋找感興趣的蛋白質結構並載下來
P.S. .pdb檔包含相當多資訊,包含結構資訊及序列資訊等
載下來後用pymol開啟該檔便可看到基本的結構了!!
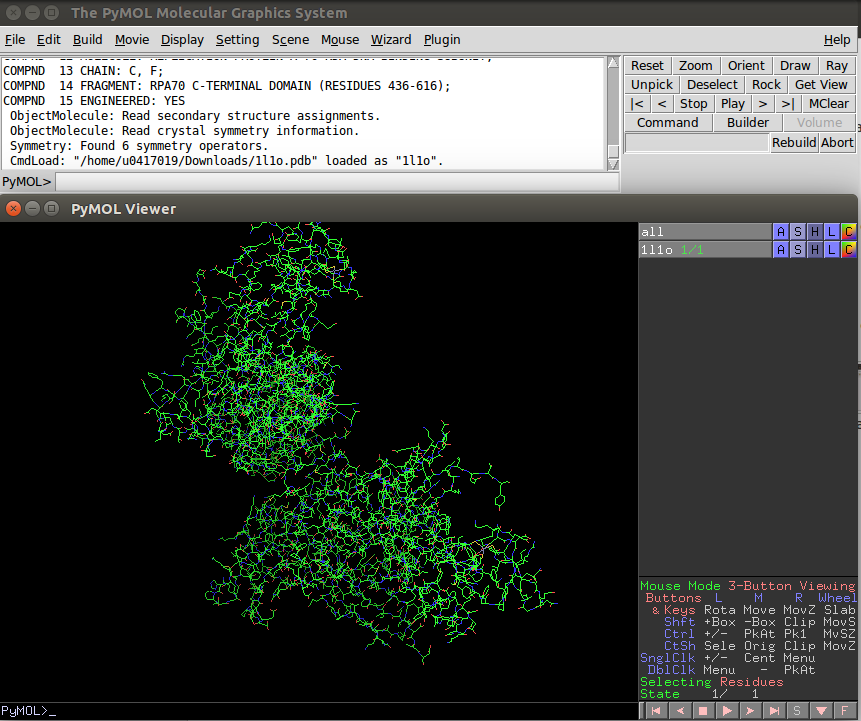
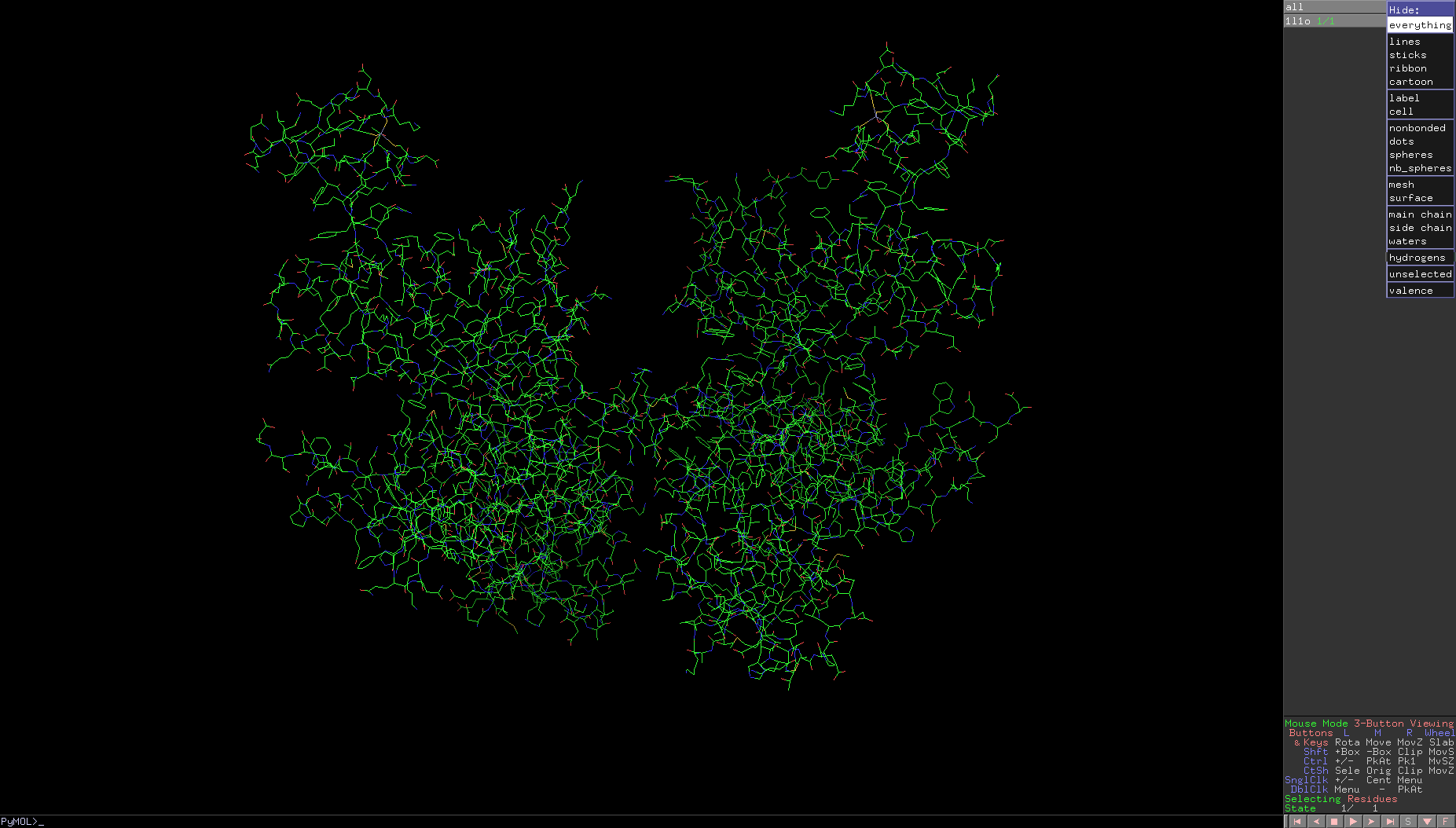
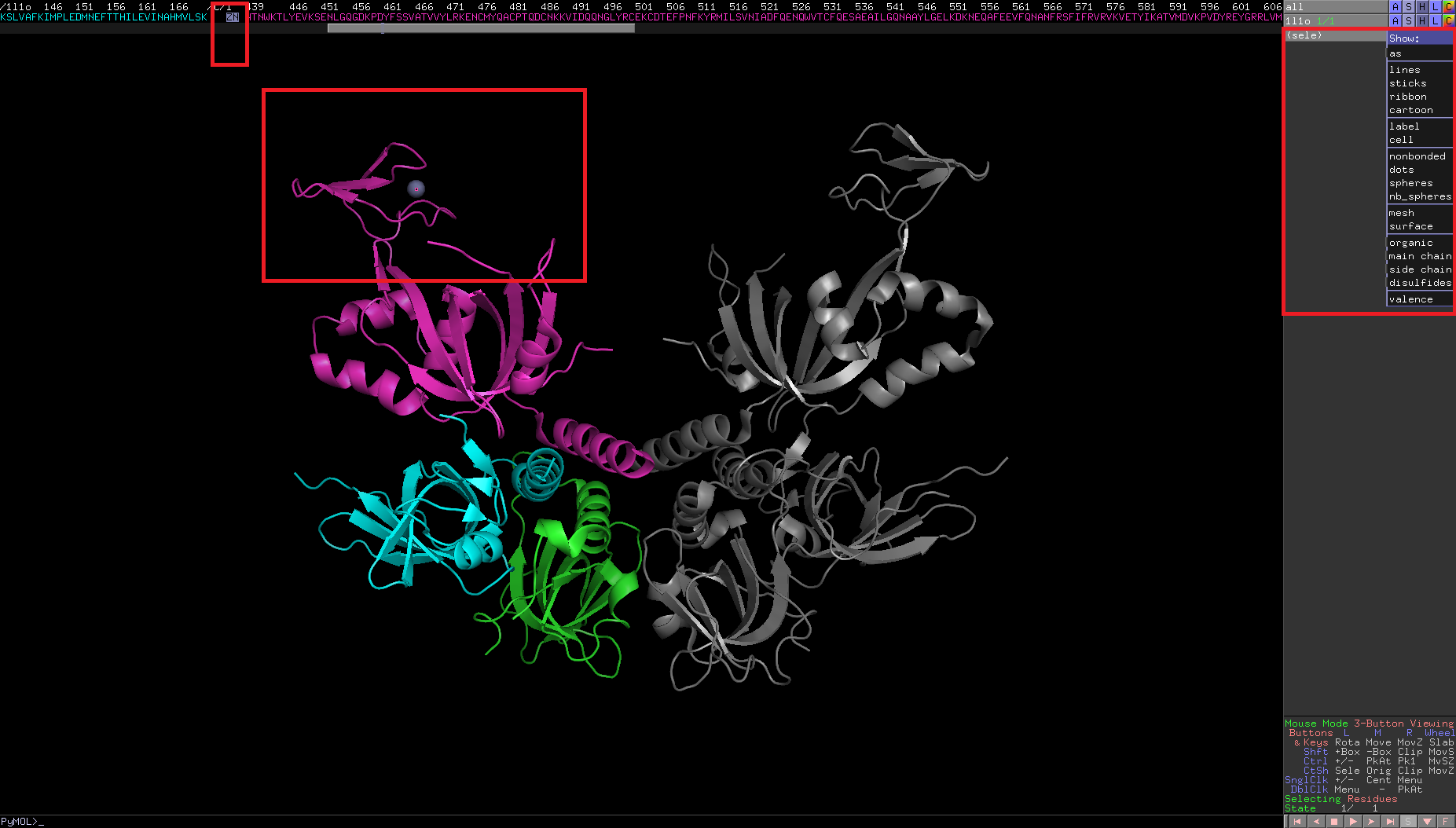
基本會用到的功能都如下圖紅框所示
A=Action
S=Show
H=Hide
L=Label
C=Color
而在操作上我們習慣以Cartoon形式來表達蛋白質的結構
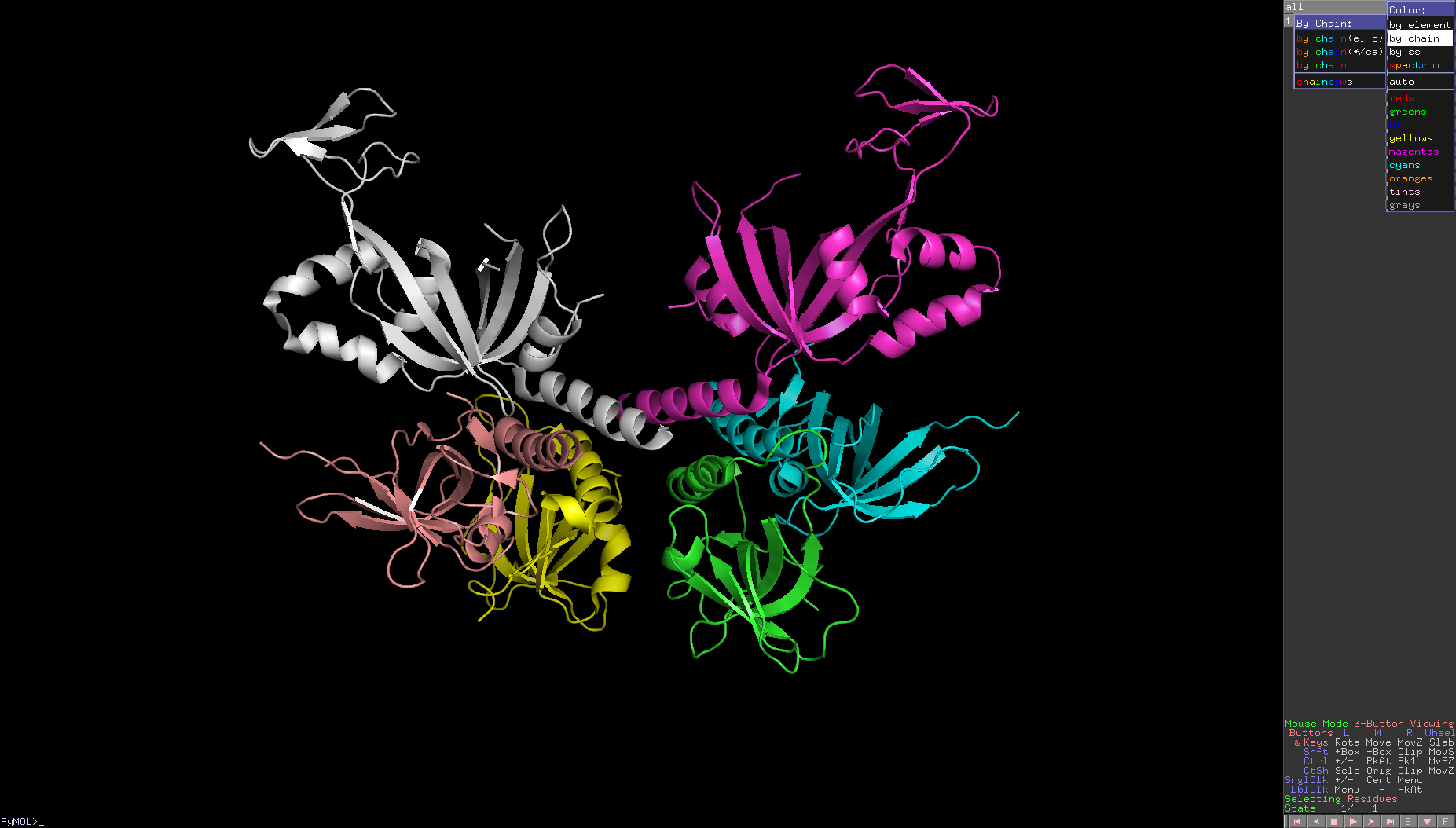
而在顏色上我們則習慣以"Color by chains"表達,因不同的chain代表不同subunit,故才有生化意義
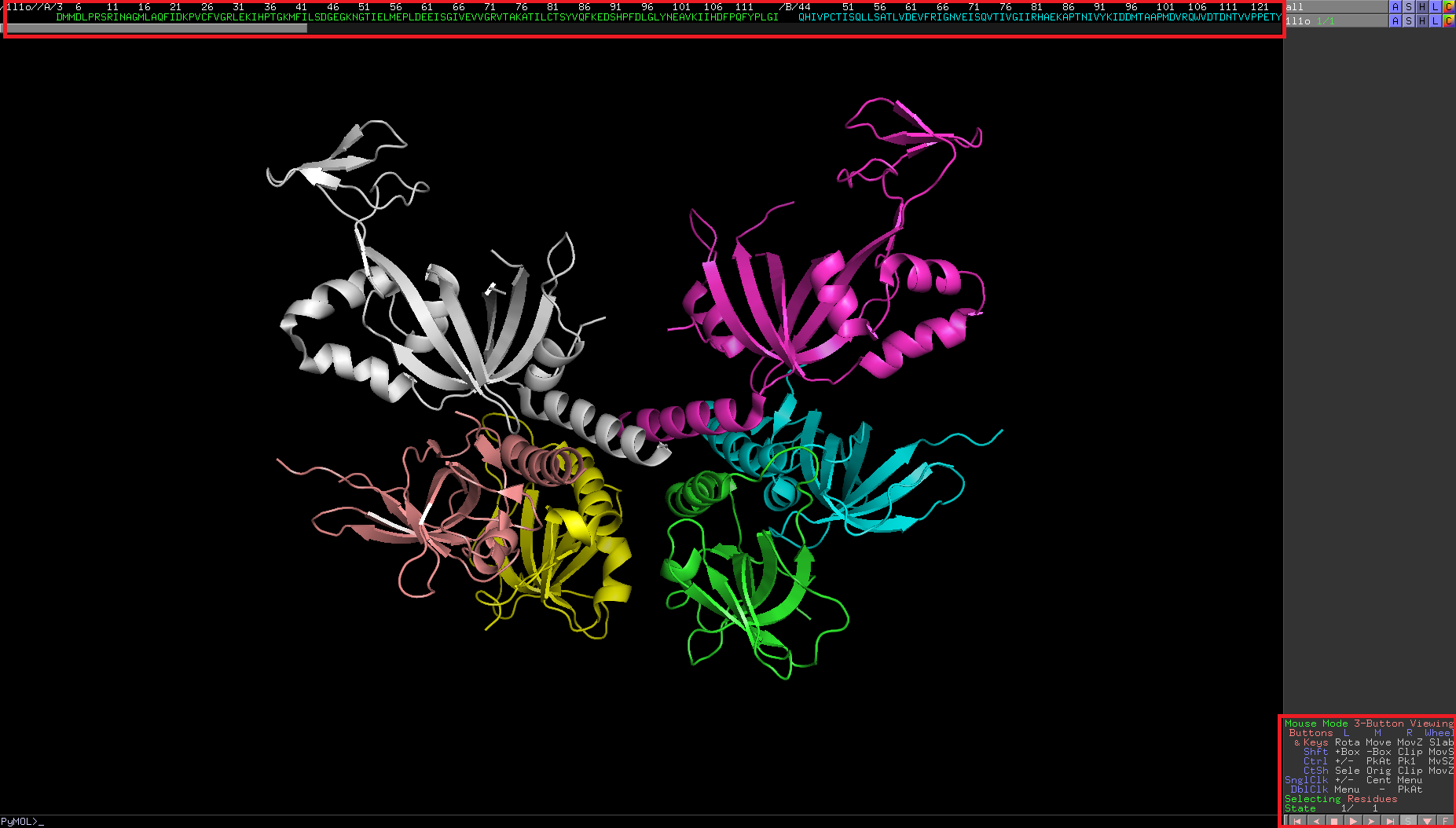
可以看到在右下角點開Sequence後上方便會跳出該蛋白質的序列,且以剛剛的配色模式幫我們搭配好了!!
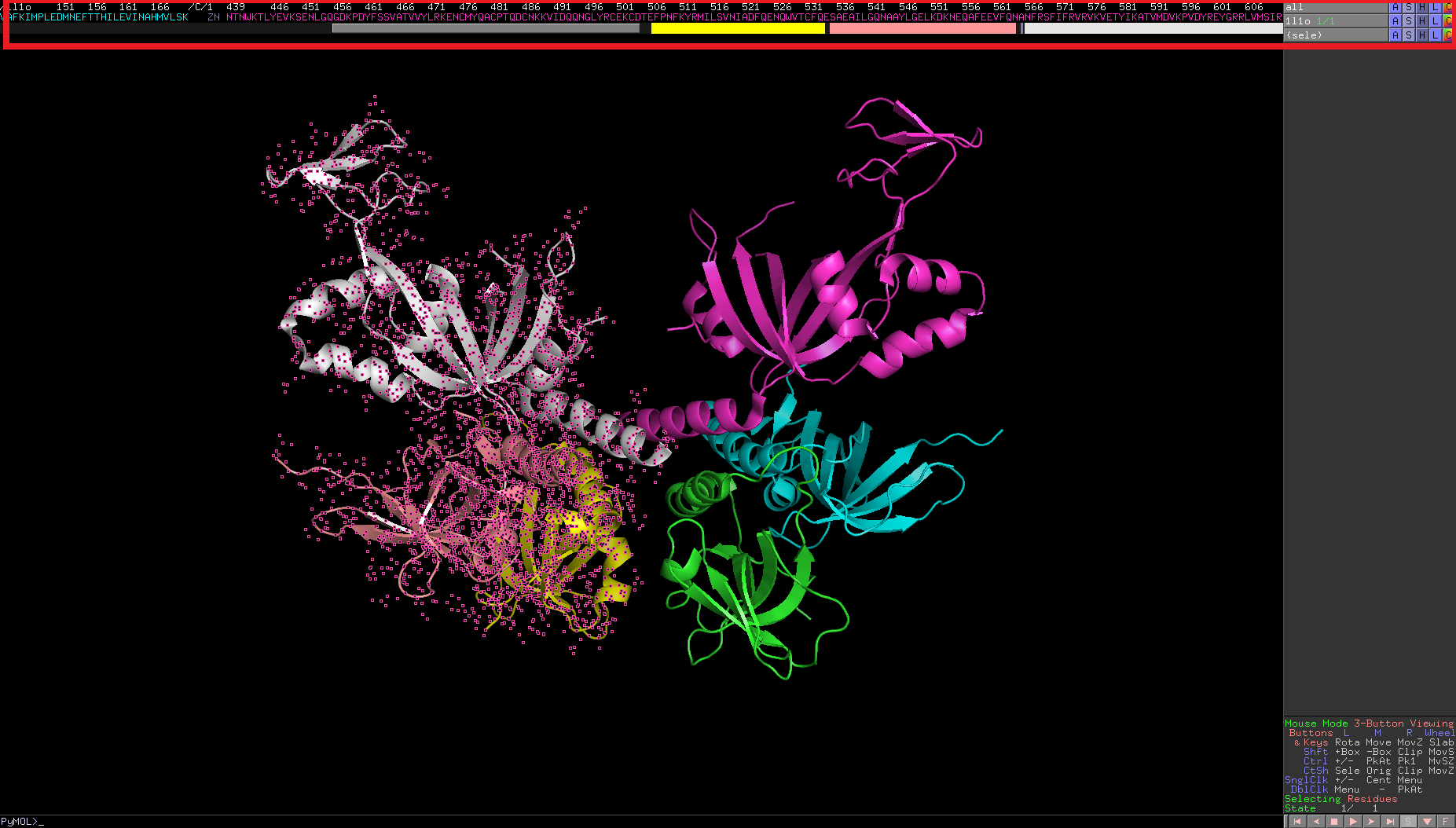
亦可直接對有興趣的序列選取後動作
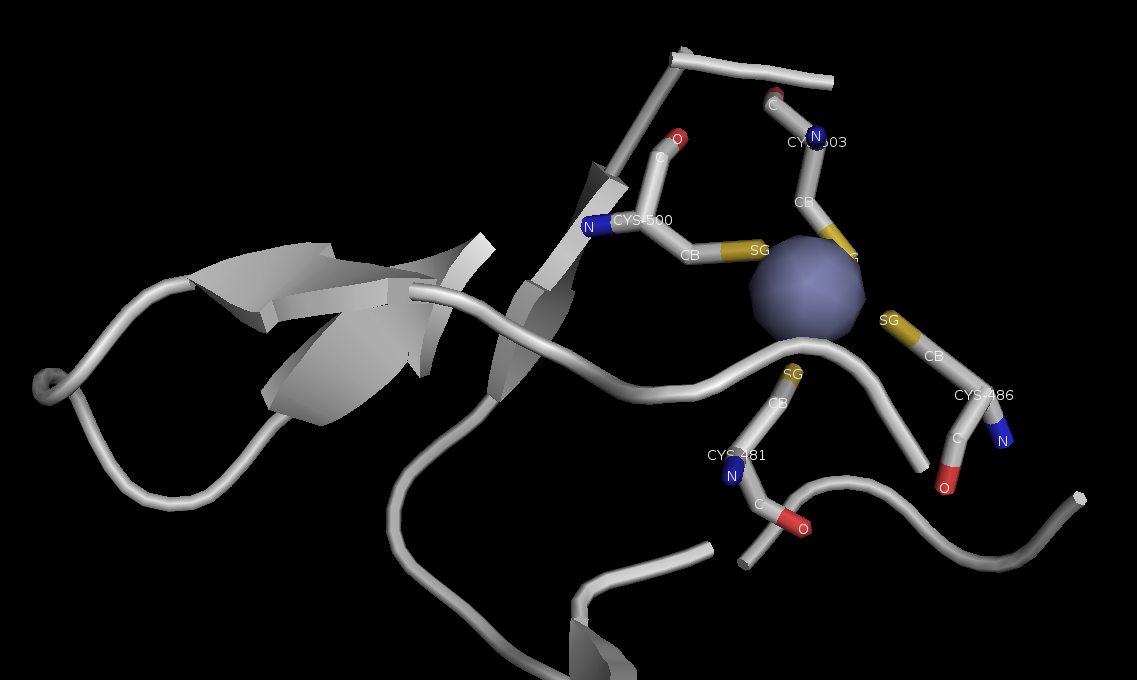
因為trimer of dimer,故將另一半用灰色表示以免混淆視聽
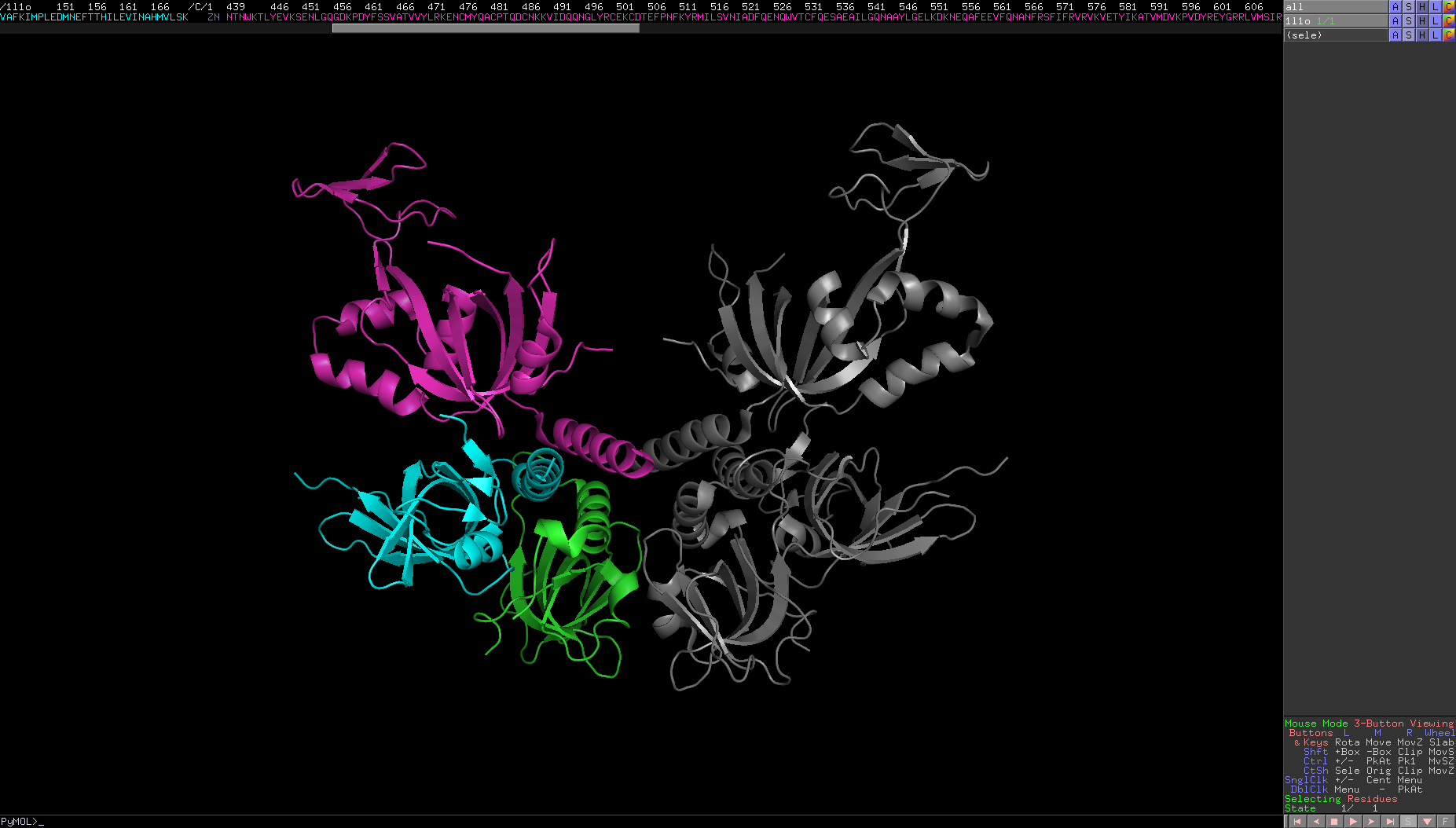
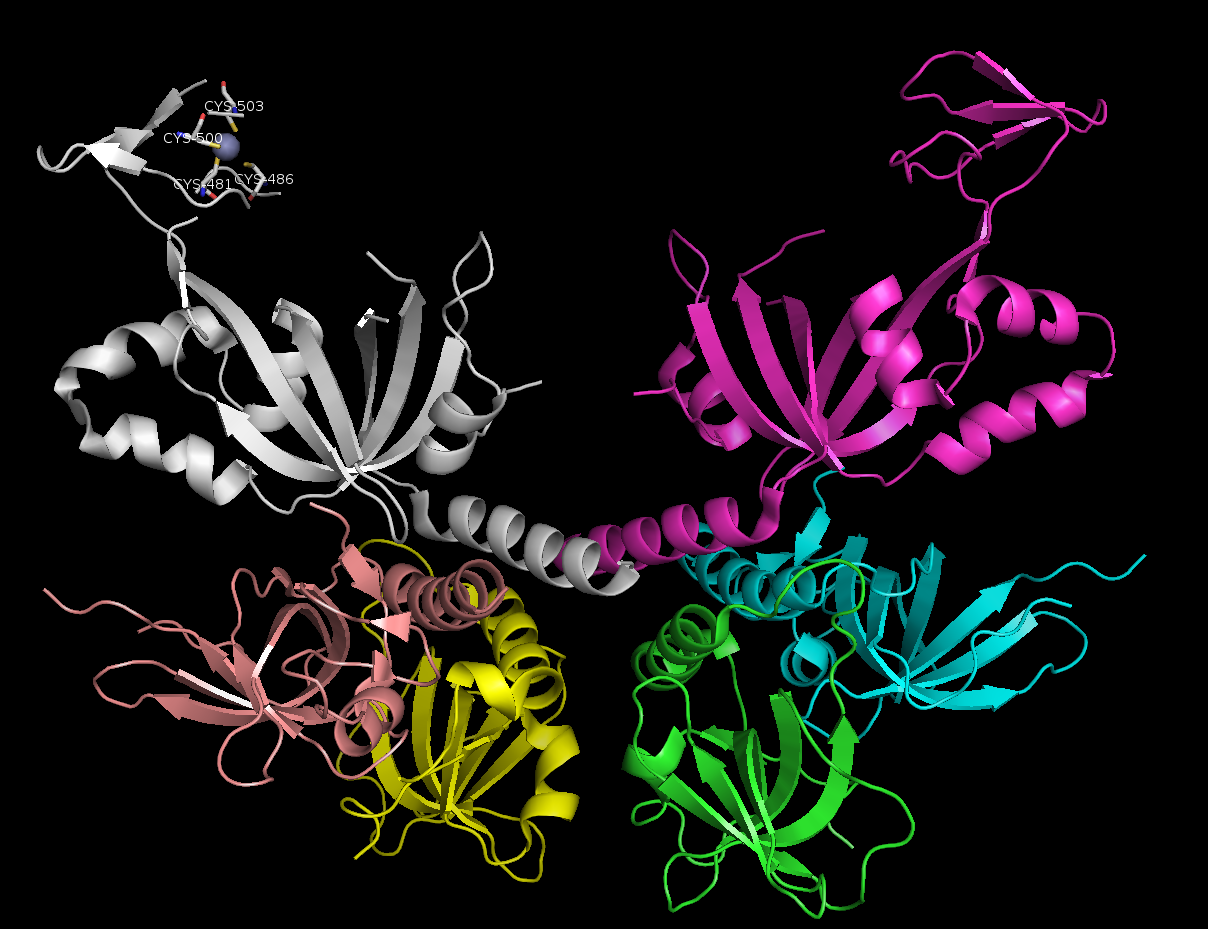
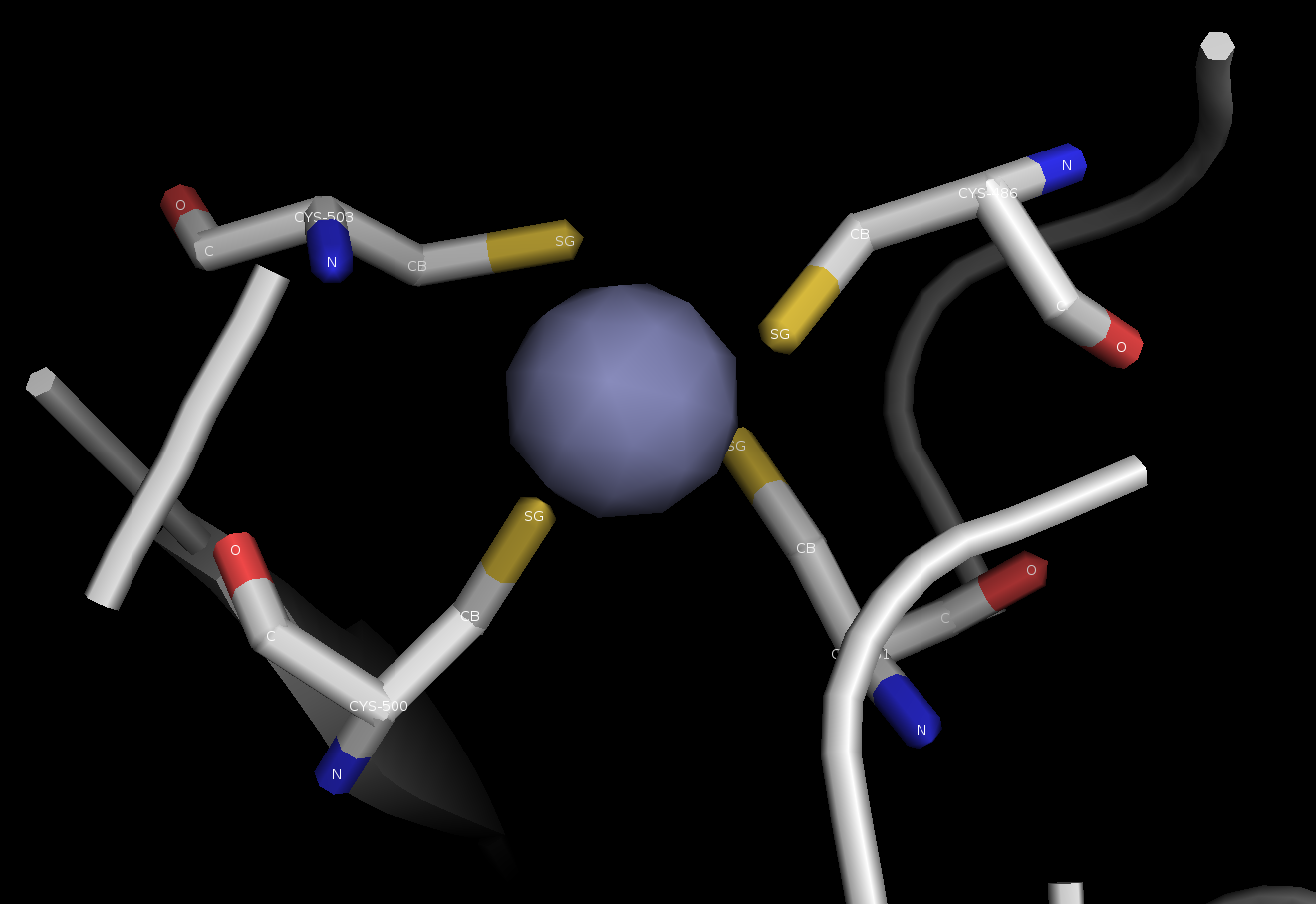
此Complex有Zn2+離子,故特別用Spheres的模式表達出來以凸顯重點
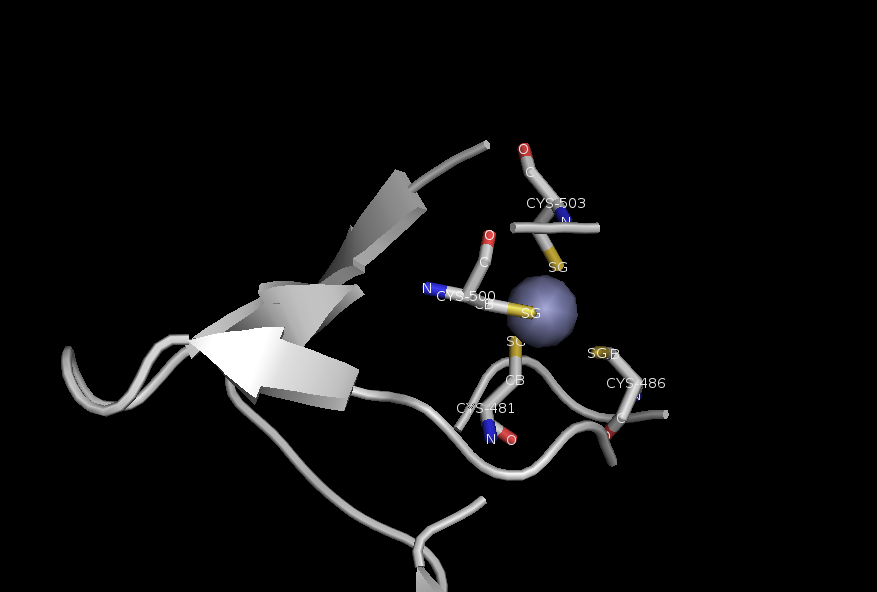
此處的motif為Zn Ribbon motif
可以看到Zn離子被4個Cysteine Coordinated著,因該四個胺基酸上有硫原子
P.S. Residues還有原子名稱皆有用Label功能標示起來
二. 去背時間!!!
1.先把背景弄白
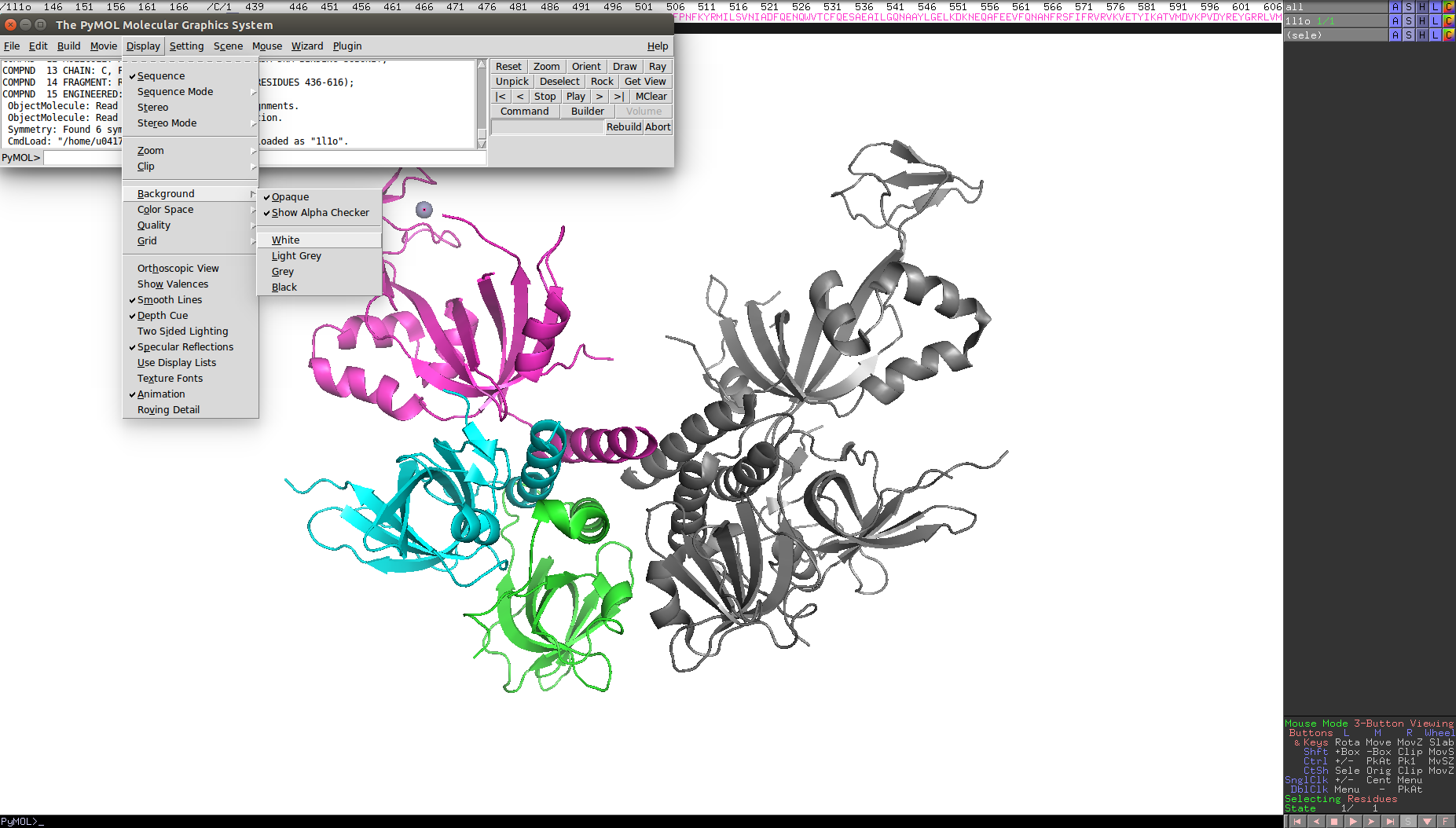
2.右鍵選取ray以消除圖案鋸齒狀醜醜的部分
3.存檔時記得將Opaque的打勾取消,再存檔便可以得到完美去背處理過的結構圖了!!

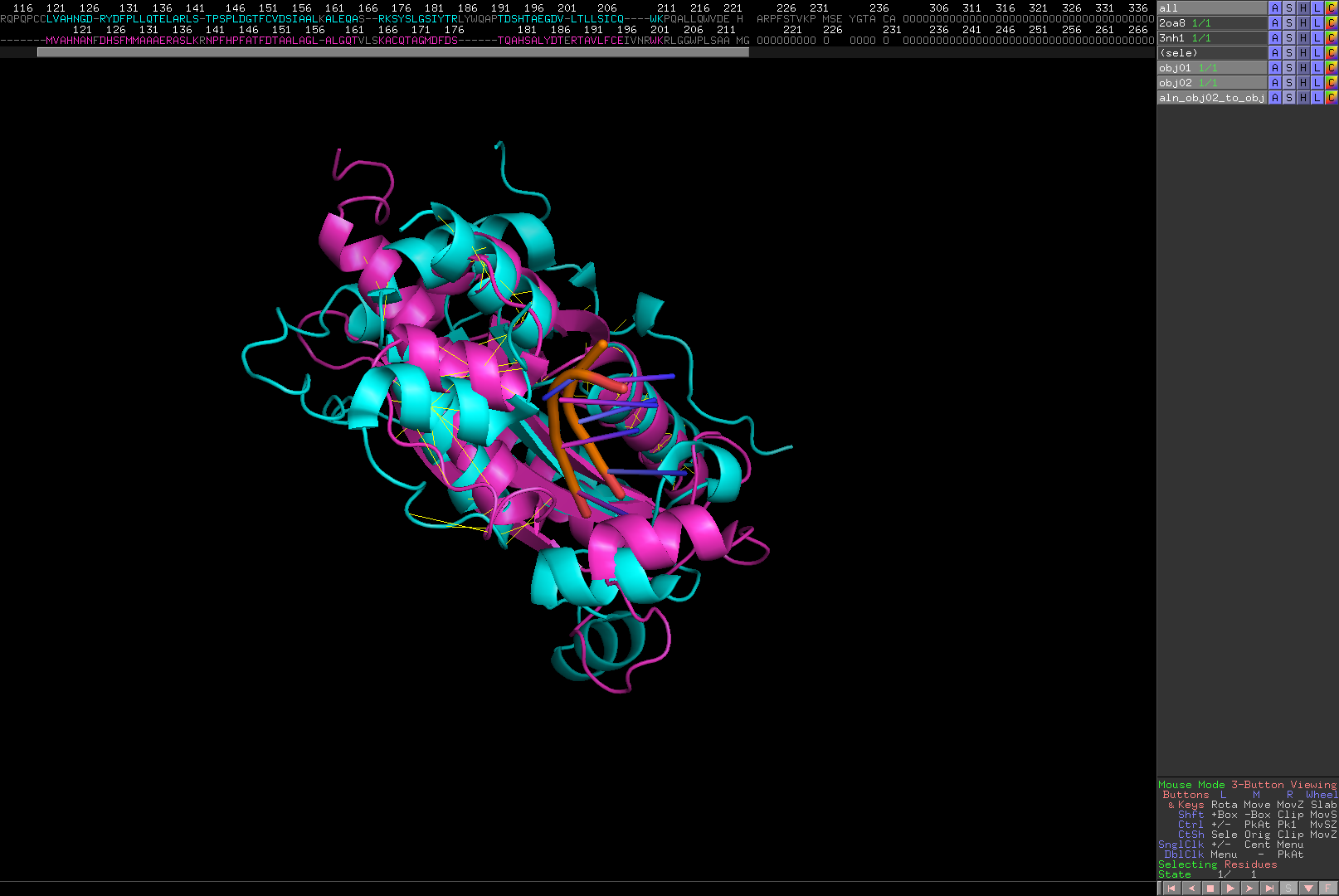
三. Structure-based alignment !!!!
找到感興趣想要拿來alignment的兩個pdb檔,以相同方式讀取便會如下圖所示
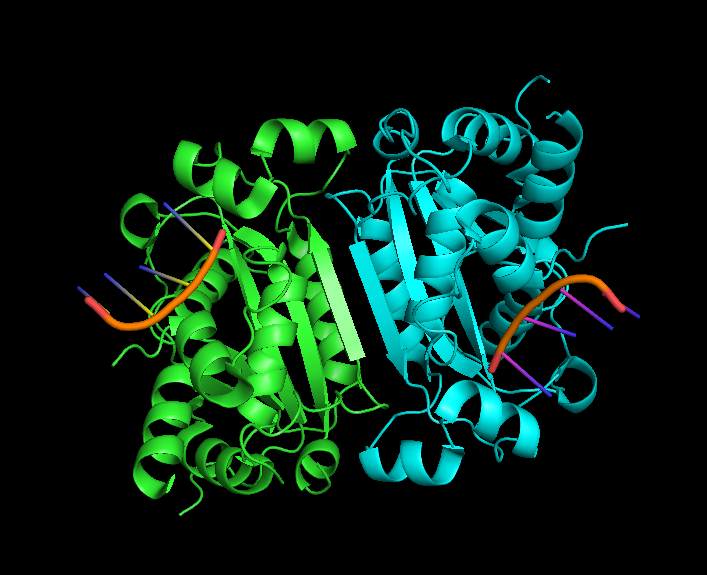
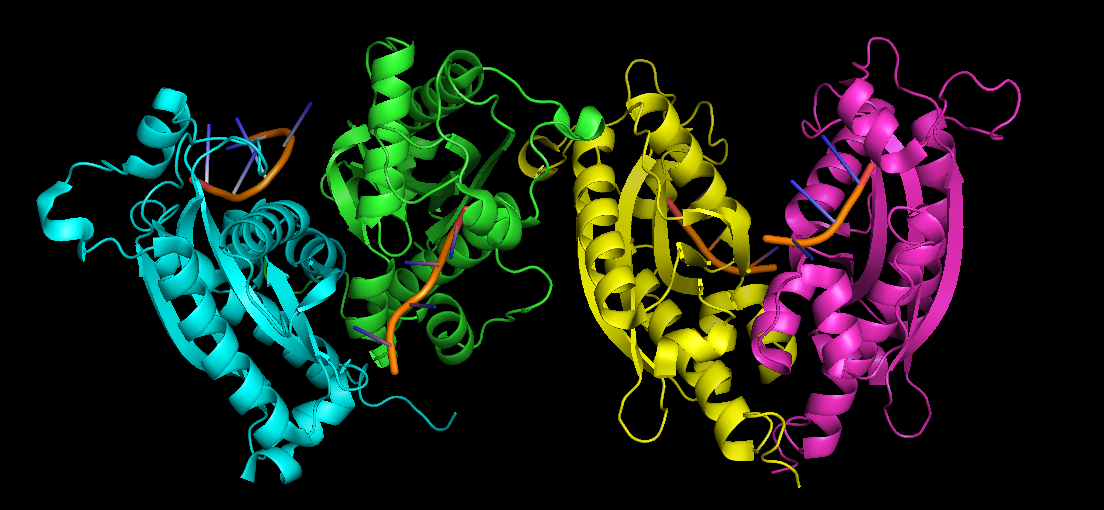
養成良好習慣!!!
避免動到原檔及操作上的機動性,故應選取有興趣的序列,在Action裡面選取Copy(duplicate) Object的動作
且通常為了提升Structure-based alignment的精確度,我們會省略掉不必要的domain和dimer or trimer等的形式而改以Monomer的形式為單一object進行alignment
P.S. 此舉將會使剛剛選取的片段成為新的物件!!
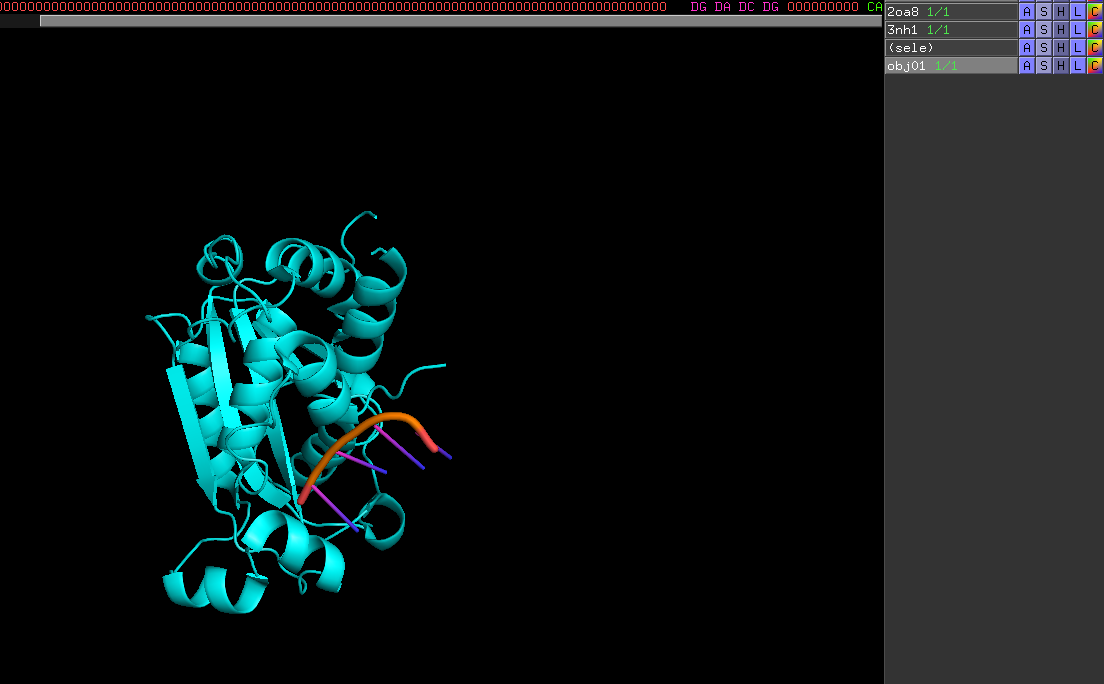
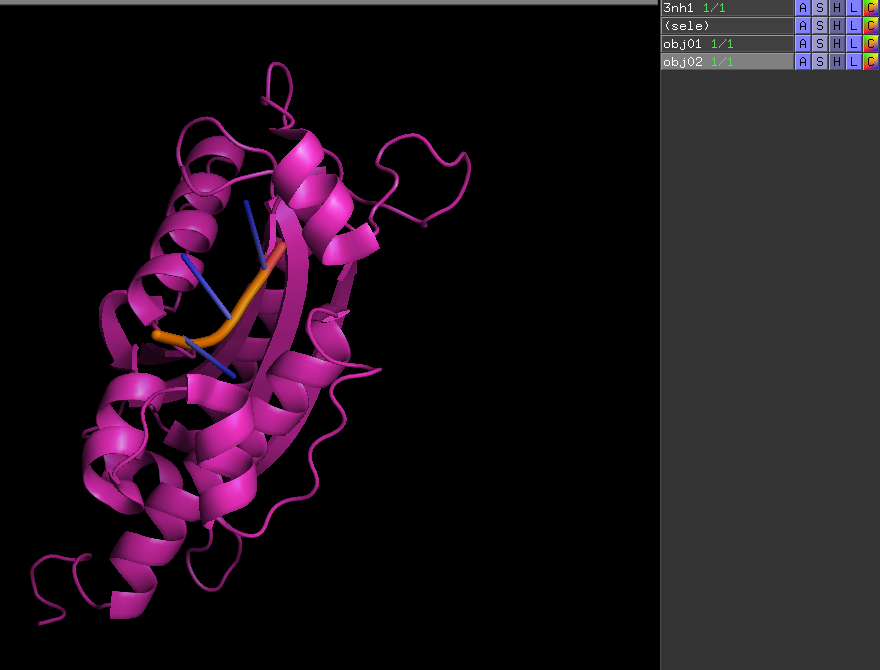
Align to molecule !!!
P.S. 記得使用剛剛新產生的兩個Objects哦 !!!
Do Re Mi So ~~~ 結構上的alignment產生!!!
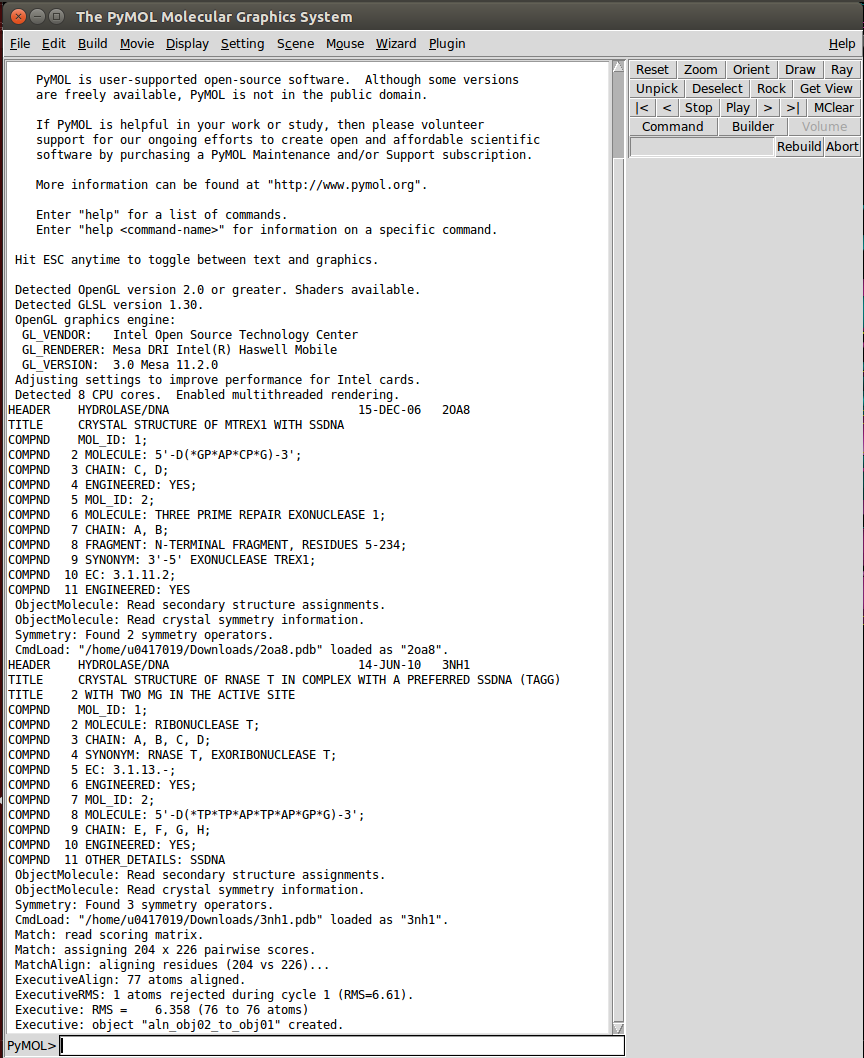
也可以在Command視窗看到RMS值哦!!!

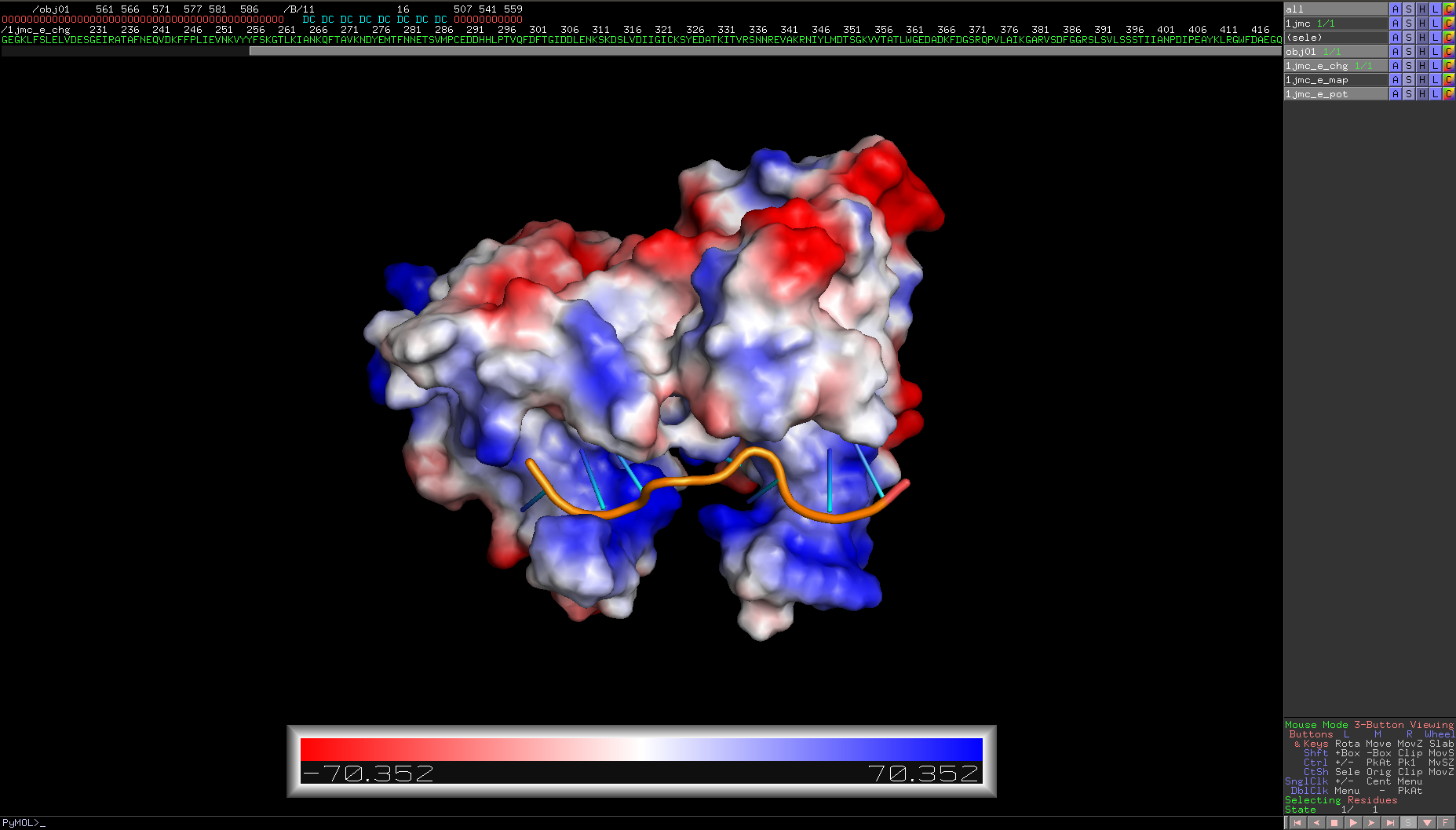
四. 表面靜電荷圖 !!!
有助於我們了解表面的電性分配如何!!
操作如下: Action -> generate -> vacuum electrostatics
P.S. 仍然是選取我們想看的片段即可
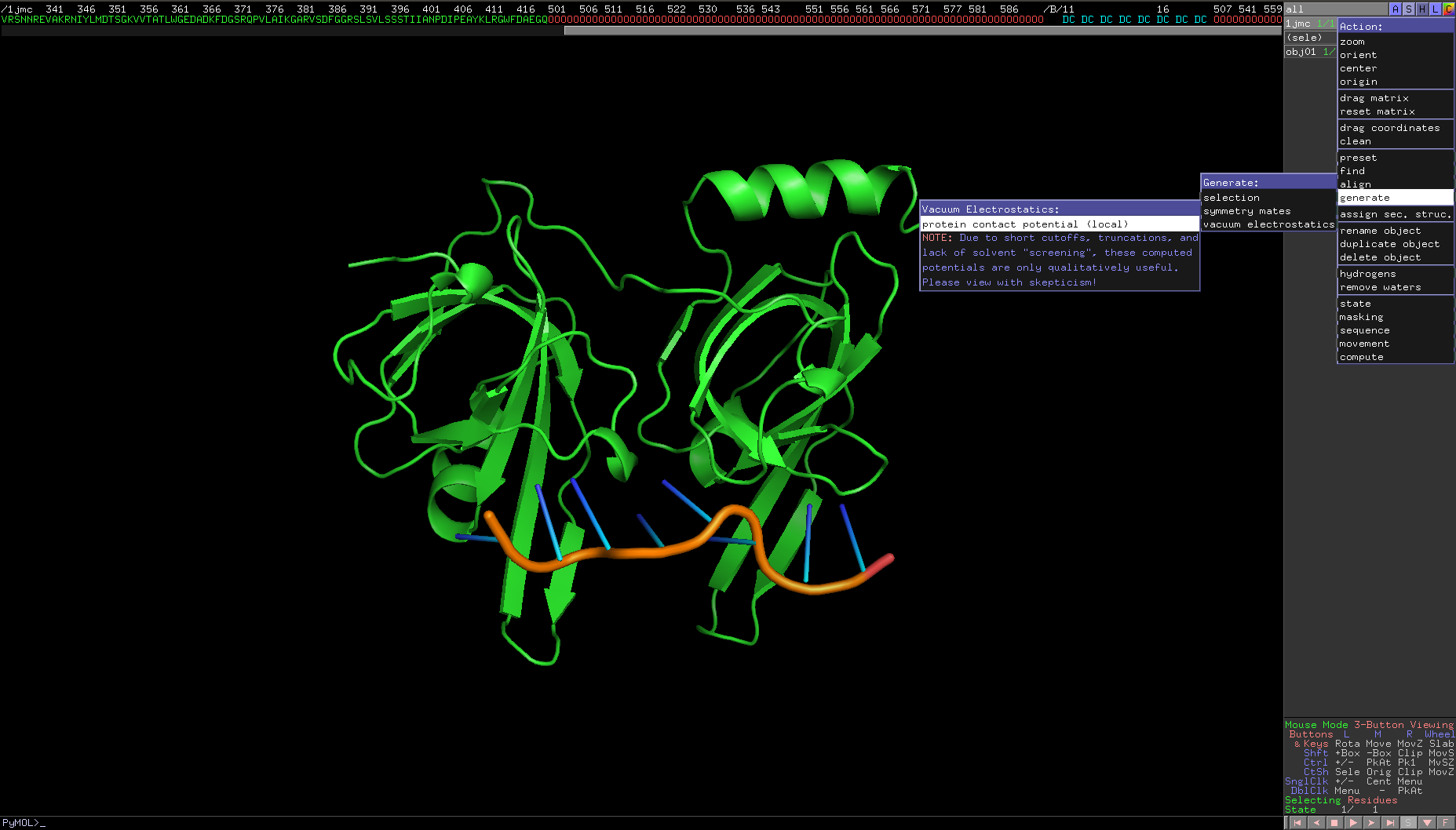
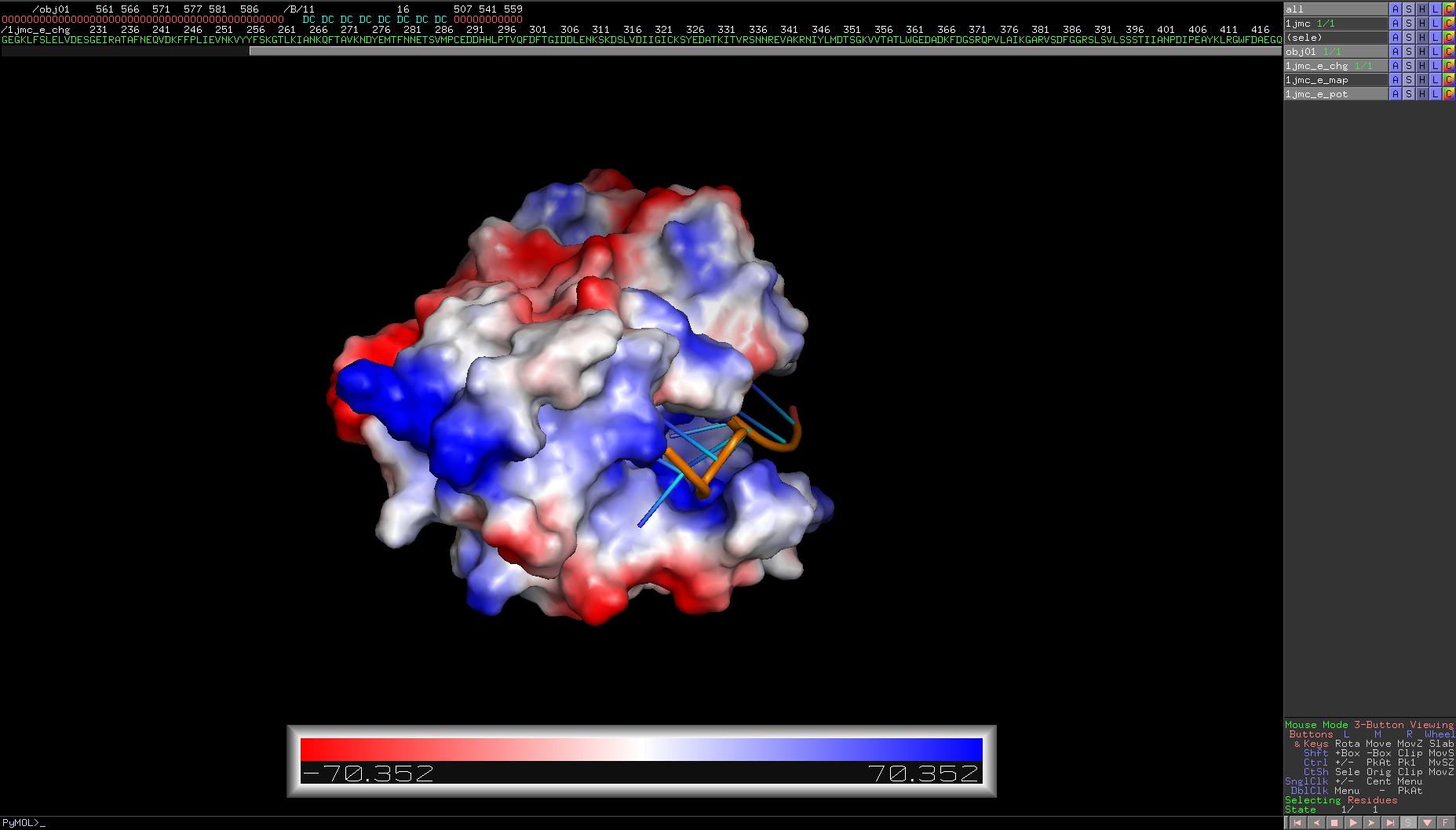
此處例子為DNA-Protein Complex,故只選取蛋白質的部分做表面靜電荷圖 !!!
可以看到DNA完美地fit蛋白質binding site的narrow groove裡 !!!
且更強烈的證據顯示,該Complex與DNA結合附近的residues呈現帶正電的藍色,與核酸(負電)的strong interaction便不證自明了 !!!
五. 暫存資料
如果遇到好不容易計算了一大堆數據,已經標示好很多東西,或是做了很多動作但卻不想一次弄完,該怎麼辦?
這時可以按照以下方法來暫存上述所有動作,待下次開啟新儲存的.pse檔即可繼續完成工作
File -> Save Session As... -> 打上檔名存成.pse的格式即可完成
六. 參考資料: 自身專題經驗
































